Questo documento mostra come iniziare a utilizzare la data science su larga scala con R su Google Cloud. È rivolto a chi ha una certa esperienza con R e con i notebook Jupyter e che ha dimestichezza con SQL.
Questo documento si concentra sull'esecuzione di analisi esplorative dei dati utilizzando istanze di Vertex AI Workbench e BigQuery. Puoi trovare il codice aggiuntivo in un blocco note Jupyter disponibile su GitHub.
Panoramica
R è uno dei linguaggi di programmazione più utilizzati per la modellistica statistica. Ha una community ampia e attiva di data scientist e professionisti del machine learning (ML). Con oltre 20.000 pacchetti nel repository open source del Comprehensive R Archive Network (CRAN), R offre strumenti per tutte le applicazioni di analisi dei dati statistici, ML e visualizzazione. R ha registrato una crescita costante negli ultimi due decenni grazie alla sua sintassi espressiva e alla completezza delle sue librerie di dati e ML.
In qualità di data scientist, potresti voler sapere come utilizzare le tue competenze con R e come sfruttare i vantaggi dei servizi cloud completamente gestiti e scalabili per la data science.
Architettura
In questa procedura dettagliata, utilizzerai istanze Vertex AI Workbench come ambienti di data science per eseguire l'analisi esplorativa dei dati (EDA). Puoi utilizzare R sui dati estratti in questa procedura dettagliata da BigQuery, il data warehouse su cloud serverless, a scalabilità elevata e conveniente di Google. Dopo averli analizzati ed elaborati, i dati trasformati vengono archiviati in Cloud Storage per ulteriori potenziali attività di ML. Questo flusso è mostrato nel seguente diagramma:

Dati di esempio
I dati di esempio per questo documento sono il
set di dati BigQuery sui viaggi in taxi di New York.
Questo set di dati pubblico include informazioni su milioni di corse in taxi che si svolgono ogni anno a New York. In questo documento utilizzi i dati del 2022, che si trovano nella tabella bigquery-public-data.new_york_taxi_trips.tlc_yellow_trips_2022 in BigQuery.
Questo documento si concentra sull'analisi esplorativa dei dati e sulla visualizzazione utilizzando R e BigQuery. I passaggi descritti in questo documento ti consentono di creare un obiettivo di ML per prevedere l'importo della tariffa del taxi (l'importo al netto di tasse, commissioni e altri extra), in base a una serie di fattori relativi alla corsa. La creazione effettiva del modello non è trattata in questo documento.
Vertex AI Workbench
Vertex AI Workbench è un servizio che offre un ambiente integrato JupyterLab con le seguenti funzionalità:
- Deployment con un solo clic. Con un solo clic puoi avviare un'istanza JupyterLab preconfigurata con i più recenti framework di machine learning e data science.
- Scalabilità on demand. Puoi iniziare con una configurazione di una piccola macchina (ad esempio 4 vCPU e 16 GB di RAM, come in questo documento) e, quando i dati diventano troppo grandi per una macchina, puoi eseguire lo scaling up aggiungendo CPU, RAM e GPU.
- Google Cloud integrazione. Le istanze di Vertex AI Workbench sono integrate con Google Cloud servizi come BigQuery. Questa integrazione semplifica il passaggio dall'importazione dei dati alla pre-elaborazione e all'esplorazione.
- Prezzi a consumo. Non sono previste tariffe minime o impegni iniziali. Per informazioni, consulta la pagina relativa ai prezzi di Vertex AI Workbench. Paghi anche per le Google Cloud risorse che utilizzi all'interno dei notebook (ad esempio BigQuery e Cloud Storage).
I notebook delle istanze Vertex AI Workbench vengono eseguiti su Deep Learning VM Image. Questo documento supporta la creazione di un'istanza di Vertex AI Workbench con R 4.3.
Utilizzare BigQuery con R
BigQuery non richiede la gestione dell'infrastruttura, quindi puoi concentrarti sull'individuazione di informazioni significative. Puoi analizzare grandi quantità di dati su larga scala e preparare set di dati per il machine learning utilizzando le ampie funzionalità di analisi SQL di BigQuery.
Per eseguire query sui dati di BigQuery utilizzando R, puoi utilizzare bigrquery, una libreria R open source. Il pacchetto bigrquery fornisce i seguenti livelli di rappresentazione astratta su BigQuery:
- L'API di basso livello fornisce wrapper sottili sull'API REST BigQuery sottostante.
- L'interfaccia DBI imbatte l'API di basso livello e rende l'utilizzo di BigQuery simile a quello di qualsiasi altro sistema di database. Questo è il livello più comodo se vuoi eseguire query SQL in BigQuery o caricare meno di 100 MB.
- L'interfaccia di dbplyr consente di trattare le tabelle BigQuery come frame di dati in memoria. Questo è il livello più pratico se non vuoi scrivere SQL, ma vuoi che sia dbplyr a farlo per te.
Questo documento utilizza l'API di basso livello di bigrquery, senza richiedere DBI o dbplyr.
Obiettivi
- Crea un'istanza di Vertex AI Workbench con il supporto di R.
- Esegui query e analizza i dati di BigQuery utilizzando la libreria R bigrquery.
- Prepara e archivia i dati per l'apprendimento automatico in Cloud Storage.
Costi
In questo documento utilizzi i seguenti componenti fatturabili di Google Cloud:
- BigQuery
- Vertex AI Workbench instances. You are also charged for resources used within notebooks, including compute resources, BigQuery, and API requests.
- Cloud Storage
Per generare una stima dei costi in base all'utilizzo previsto,
utilizza il Calcolatore prezzi.
Prima di iniziare
- Sign in to your Google Cloud account. If you're new to Google Cloud, create an account to evaluate how our products perform in real-world scenarios. New customers also get $300 in free credits to run, test, and deploy workloads.
-
In the Google Cloud console, on the project selector page, select or create a Google Cloud project.
-
Make sure that billing is enabled for your Google Cloud project.
-
Enable the Compute Engine API.
-
In the Google Cloud console, on the project selector page, select or create a Google Cloud project.
-
Make sure that billing is enabled for your Google Cloud project.
-
Enable the Compute Engine API.
Crea un'istanza di Vertex AI Workbench
Il primo passaggio consiste nel creare un'istanza di Vertex AI Workbench che puoi utilizzare per questa procedura dettagliata.
Nella console Google Cloud, vai alla pagina Workbench.
Nella scheda Istanze, fai clic su Crea nuova.
Nella finestra Nuova istanza, fai clic su Crea. Per questa procedura dettagliata, mantieni tutti i valori predefiniti.
L'avvio dell'istanza di Vertex AI Workbench può richiedere 2-3 minuti. Al termine, l'istanza viene elencata automaticamente nel riquadro Istanze di notebook e accanto al nome dell'istanza viene visualizzato un link Apri JupyterLab. Se il link per aprire JupyterLab non viene visualizzato nell'elenco dopo alcuni minuti, aggiorna la pagina.
Apri JupyterLab e installa R
Per completare la procedura dettagliata nel notebook, devi aprire l'ambiente JupyterLab, installare R, clonare il repository GitHub vertex-ai-samples e aprire il notebook.
Nell'elenco delle istanze, fai clic su Apri JupyterLab. Viene aperto l'ambiente JupyterLab in un'altra scheda del browser.
Nell'ambiente JupyterLab, fai clic su Nuovo Avvio app e poi su Terminale nella scheda Avvio app.
Nel riquadro del terminale, installa R:
conda create -n r conda activate r conda install -c r r-essentials r-base=4.3.2Durante l'installazione, ogni volta che ti viene chiesto di continuare, digita
y. Il completamento dell'installazione potrebbe richiedere alcuni minuti. Al termine dell'installazione, l'output è simile al seguente:done Executing transaction: done ® jupyter@instance-INSTANCE_NUMBER:~$dove INSTANCE_NUMBER è il numero univoco assegnato alla tua istanza di Vertex AI Workbench.
Al termine dell'esecuzione dei comandi nel terminale, aggiorna la pagina del browser e apri Avvio app facendo clic su Nuovo Avvio app.
La scheda Avvio mostra le opzioni per avviare R in un notebook o nella console e per creare un file R.
Fai clic sulla scheda Terminale, quindi clona il repository GitHub vertex-ai-samples:
git clone https://github.com/GoogleCloudPlatform/vertex-ai-samples.gitAl termine del comando, vedrai la cartella
vertex-ai-samplesnel riquadro del browser di file dell'ambiente JupyterLab.Nel browser di file, apri
vertex-ai-samples>notebooks>community>exploratory_data_analysis. Vedrai ileda_with_r_and_bigquery.ipynbblocco note.
Apri il notebook e configura R
Nel browser di file, apri il notebook
eda_with_r_and_bigquery.ipynb.Questo notebook illustra l'analisi esplorativa dei dati con R e BigQuery. Nel resto di questo documento, lavorerai nel blocco note ed eseguirai il codice che vedi al suo interno.
Controlla la versione di R utilizzata dal notebook:
versionNel campo
version.stringdell'output dovrebbe essere visualizzatoR version 4.3.2, che hai installato nella sezione precedente.Cerca e installa i pacchetti R necessari se non sono già disponibili nella sessione corrente:
# List the necessary packages needed_packages <- c("dplyr", "ggplot2", "bigrquery") # Check if packages are installed installed_packages <- .packages(all.available = TRUE) missing_packages <- needed_packages[!(needed_packages %in% installed_packages)] # If any packages are missing, install them if (length(missing_packages) > 0) { install.packages(missing_packages) }Carica i pacchetti richiesti:
# Load the required packages lapply(needed_packages, library, character.only = TRUE)Autentica
bigrqueryutilizzando l'autenticazione lato client:bq_auth(use_oob = True)Imposta il nome del progetto che vuoi utilizzare per questo notebook sostituendo
[YOUR-PROJECT-ID]con un nome:# Set the project ID PROJECT_ID <- "[YOUR-PROJECT-ID]"Imposta il nome del bucket Cloud Storage in cui archiviare i dati di output sostituendo
[YOUR-BUCKET-NAME]con un nome univoco a livello globale:BUCKET_NAME <- "[YOUR-BUCKET-NAME]"Imposta l'altezza e la larghezza predefinite per i grafici che verranno generati più avanti nel notebook:
options(repr.plot.height = 9, repr.plot.width = 16)
Esegui query sui dati di BigQuery
In questa sezione del notebook, leggi i risultati dell'esecuzione di un statement SQL di BigQuery in R ed esamini preliminarmente i dati.
Crea un'istruzione SQL BigQuery che estrae alcuni possibili predittori e la variabile di previsione target per un campione di viaggi. La seguente query esclude alcuni valori outlier o privi di significato nei campi che vengono letti per l'analisi.
sql_query_template <- " SELECT TIMESTAMP_DIFF(dropoff_datetime, pickup_datetime, MINUTE) AS trip_time_minutes, passenger_count, ROUND(trip_distance, 1) AS trip_distance_miles, rate_code, /* Mapping from rate code to type from description column in BigQuery table schema */ (CASE WHEN rate_code = '1.0' THEN 'Standard rate' WHEN rate_code = '2.0' THEN 'JFK' WHEN rate_code = '3.0' THEN 'Newark' WHEN rate_code = '4.0' THEN 'Nassau or Westchester' WHEN rate_code = '5.0' THEN 'Negotiated fare' WHEN rate_code = '6.0' THEN 'Group ride' /* Several NULL AND some '99.0' values go here */ ELSE 'Unknown' END) AS rate_type, fare_amount, CAST(ABS(FARM_FINGERPRINT( CONCAT( CAST(trip_distance AS STRING), CAST(fare_amount AS STRING) ) )) AS STRING) AS key FROM `bigquery-public-data.new_york_taxi_trips.tlc_yellow_trips_2022` /* Filter out some outlier or hard to understand values */ WHERE (TIMESTAMP_DIFF(dropoff_datetime, pickup_datetime, MINUTE) BETWEEN 0.01 AND 120) AND (passenger_count BETWEEN 1 AND 10) AND (trip_distance BETWEEN 0.01 AND 100) AND (fare_amount BETWEEN 0.01 AND 250) LIMIT %s "La colonna
keyè un identificatore di riga generato in base ai valori concatenati delle colonnetrip_distanceefare_amount.Esegui la query e recupera gli stessi dati come tibble in memoria, che è simile a un frame di dati.
sample_size <- 10000 sql_query <- sprintf(sql_query_template, sample_size) taxi_trip_data <- bq_table_download( bq_project_query( PROJECT_ID, query = sql_query ) )Visualizza i risultati recuperati:
head(taxi_trip_data)L'output è una tabella simile alla seguente immagine:
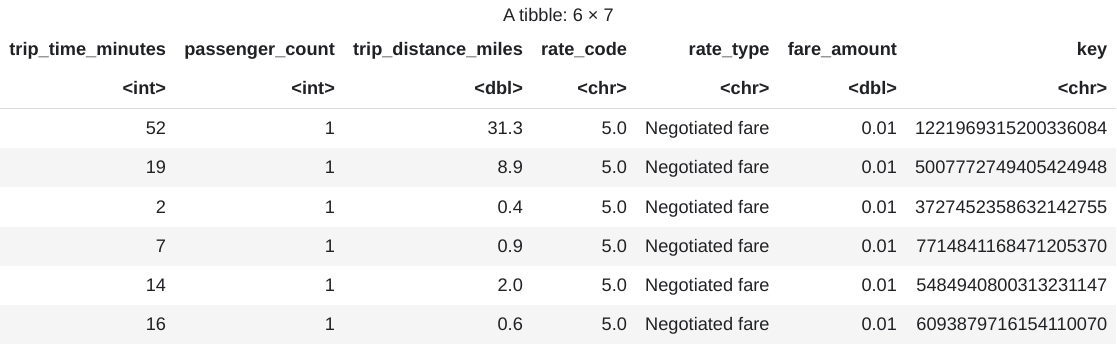
I risultati mostrano le seguenti colonne di dati sui viaggi:
- Numero intero
trip_time_minutes - Numero intero
passenger_count trip_distance_milesdoppiarate_codecarattererate_typecaratterefare_amountdoppiakeycarattere
- Numero intero
Visualizza il numero di righe e i tipi di dati di ogni colonna:
str(taxi_trip_data)L'output è simile al seguente:
tibble [10,000 x 7] (S3: tbl_df/tbl/data.frame) $ trip_time_minutes : int [1:10000] 52 19 2 7 14 16 1 2 2 6 ... $ passenger_count : int [1:10000] 1 1 1 1 1 1 1 1 3 1 ... $ trip_distance_miles: num [1:10000] 31.3 8.9 0.4 0.9 2 0.6 1.7 0.4 0.5 0.2 ... $ rate_code : chr [1:10000] "5.0" "5.0" "5.0" "5.0" ... $ rate_type : chr [1:10000] "Negotiated fare" "Negotiated fare" "Negotiated fare" "Negotiated fare" ... $ fare_amount : num [1:10000] 0.01 0.01 0.01 0.01 0.01 0.01 0.01 0.01 0.01 0.01 ... $ key : chr [1:10000] "1221969315200336084" 5007772749405424948" "3727452358632142755" "77714841168471205370" ...Visualizza un riepilogo dei dati recuperati:
summary(taxi_trip_data)L'output è simile al seguente:
trip_time_minutes passenger_count trip_distance_miles rate_code Min. : 1.00 Min. :1.000 Min. : 0.000 Length:10000 1st Qu.: 20.00 1st Qu.:1.000 1st Qu.: 3.700 Class :character Median : 24.00 Median :1.000 Median : 4.800 Mode :character Mean : 30.32 Mean :1.465 Mean : 9.639 3rd Qu.: 39.00 3rd Qu.:2.000 3rd Qu.:17.600 Max. :120.00 Max. :9.000 Max. :43.700 rate_type fare_amount key Length:10000 Min. : 0.01 Length:10000 Class :character 1st Qu.: 16.50 Class :character Mode :character Median : 16.50 Mode :character Mean : 31.22 3rd Qu.: 52.00 Max. :182.50
Visualizzare i dati utilizzando ggplot2
In questa sezione del notebook, utilizzerai la libreria ggplot2 in R per studiare alcune delle variabili del set di dati di esempio.
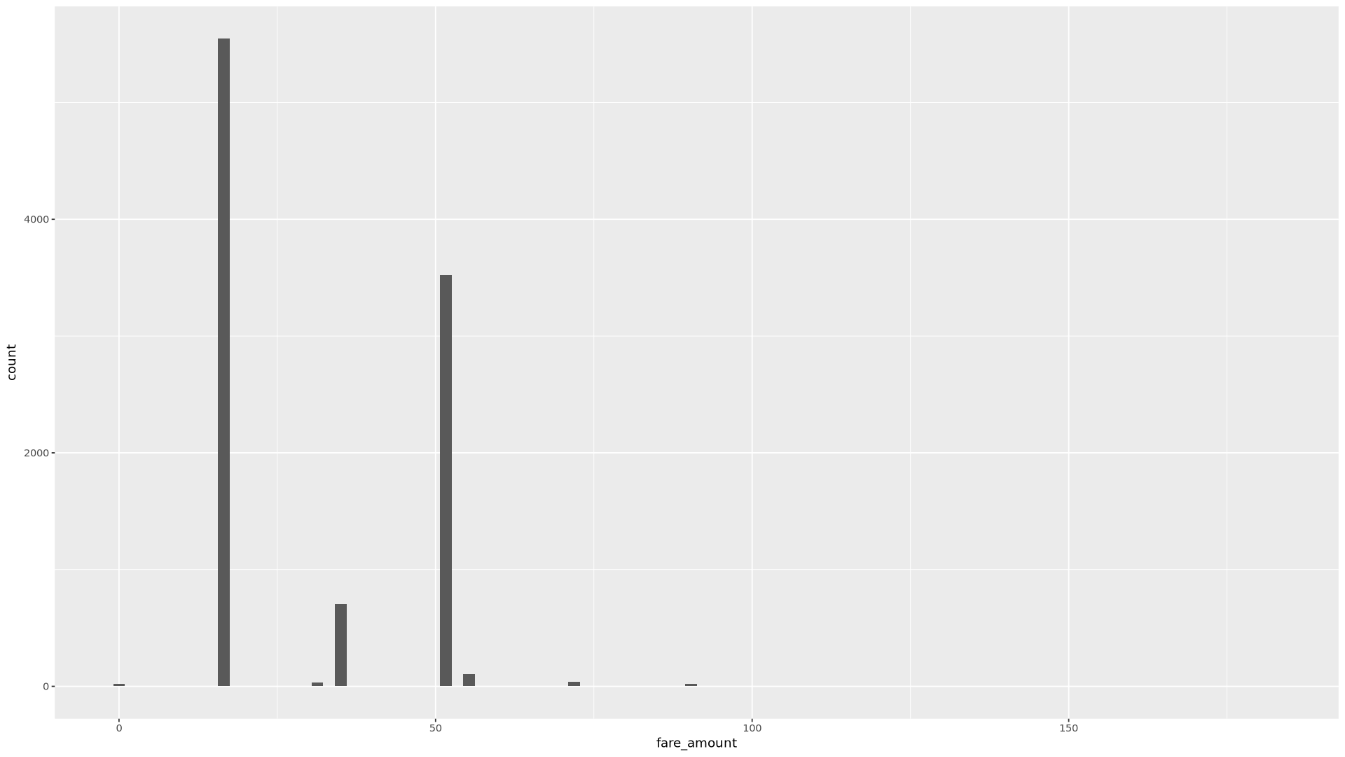
Visualizza la distribuzione dei valori
fare_amountutilizzando un istogramma:ggplot( data = taxi_trip_data, aes(x = fare_amount) ) + geom_histogram(bins = 100)Il grafico risultante è simile a quello nell'immagine seguente:
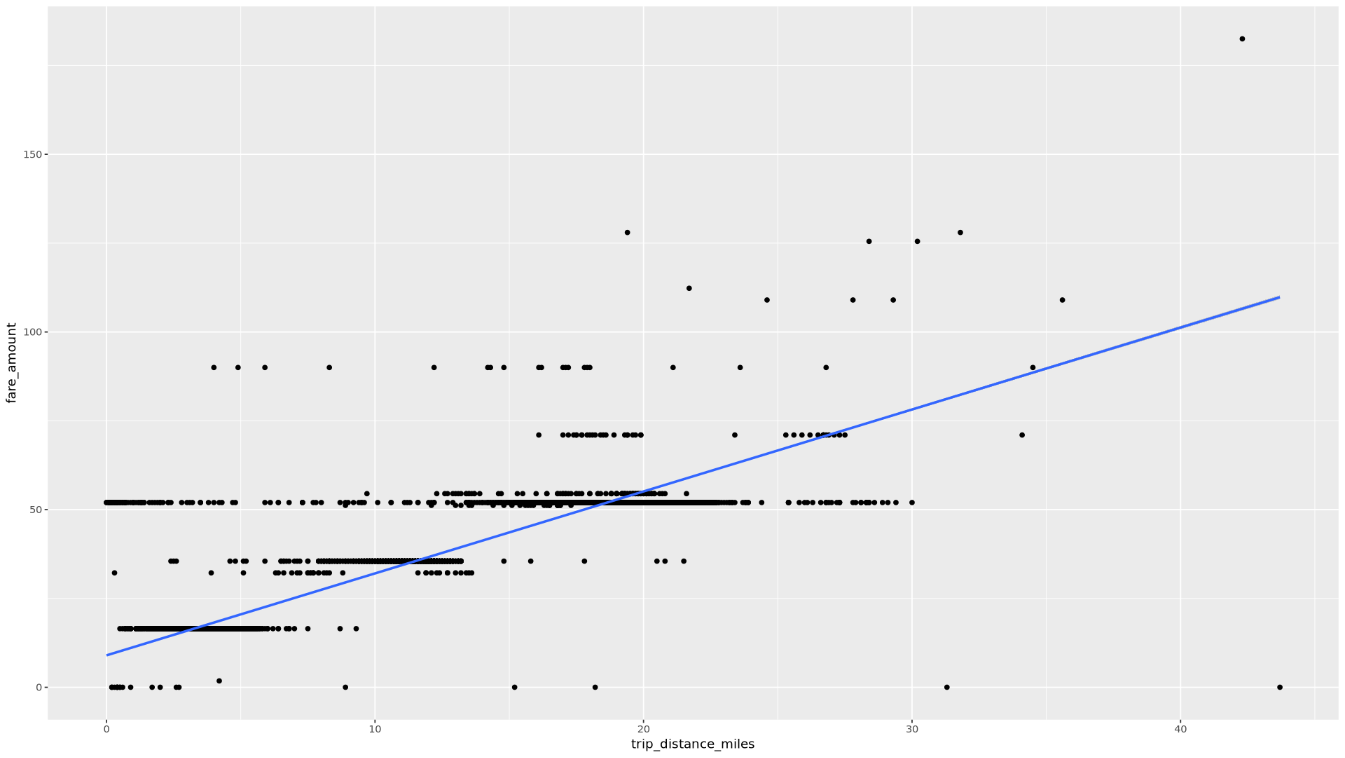
Mostra la relazione tra
trip_distanceefare_amountutilizzando un grafico a dispersione:ggplot( data = taxi_trip_data, aes(x = trip_distance_miles, y = fare_amount) ) + geom_point() + geom_smooth(method = "lm")Il grafico risultante è simile a quello nell'immagine seguente:
Elaborare i dati in BigQuery da R
Quando lavori con set di dati di grandi dimensioni, ti consigliamo di eseguire il maggior numero possibile di analisi (aggregazione, filtri, unioni, colonne di calcolo e così via) in BigQuery e poi di recuperare i risultati. L'esecuzione di queste attività in R è meno efficiente. L'utilizzo di BigQuery per l'analisi sfrutta la scalabilità e le prestazioni di BigQuery e garantisce che i risultati restituiti possano essere inseriti nella memoria in R.
Nel notebook, crea una funzione che trovi il numero di corse e l'importo medio della tariffa per ogni valore della colonna scelta:
get_distinct_value_aggregates <- function(column) { query <- paste0( 'SELECT ', column, ', COUNT(1) AS num_trips, AVG(fare_amount) AS avg_fare_amount FROM `bigquery-public-data.new_york_taxi_trips.tlc_yellow_trips_2022` WHERE (TIMESTAMP_DIFF(dropoff_datetime, pickup_datetime, MINUTE) BETWEEN 0.01 AND 120) AND (passenger_count BETWEEN 1 AND 10) AND (trip_distance BETWEEN 0.01 AND 100) AND (fare_amount BETWEEN 0.01 AND 250) GROUP BY 1 ' ) bq_table_download( bq_project_query( PROJECT_ID, query = query ) ) }Richiama la funzione utilizzando la colonna
trip_time_minutesdefinita utilizzando la funzionalità del timestamp in BigQuery:df <- get_distinct_value_aggregates( 'TIMESTAMP_DIFF(dropoff_datetime, pickup_datetime, MINUTE) AS trip_time_minutes') ggplot( data = df, aes(x = trip_time_minutes, y = num_trips) ) + geom_line() ggplot( data = df, aes(x = trip_time_minutes, y = avg_fare_amount) ) + geom_line()Il notebook mostra due grafici. Il primo grafico mostra il numero di viaggi in base alla durata in minuti. Il secondo grafico mostra l'importo medio della tariffa per numero di corse in base all'ora di partenza.
L'output del primo comando
ggplotè il seguente e mostra il numero di corse in base alla durata (in minuti):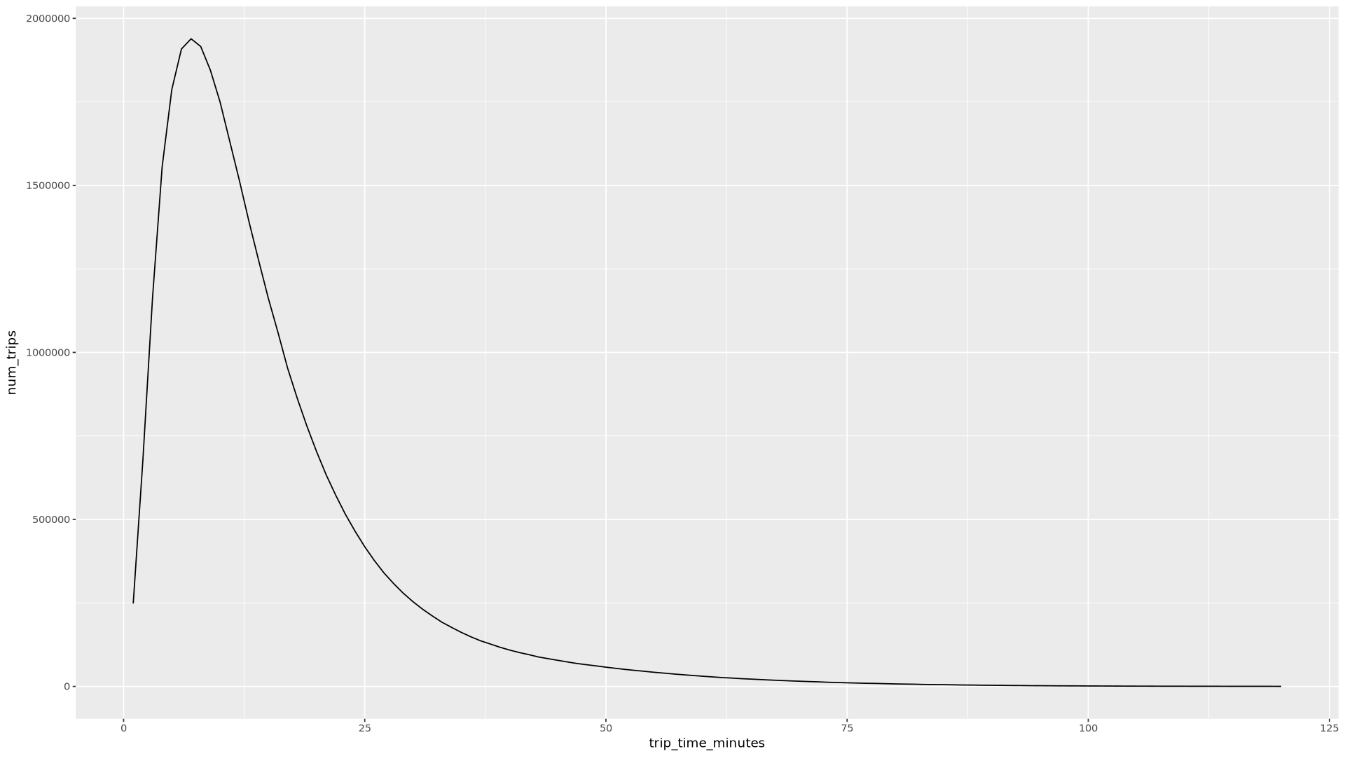
L'output del secondo comando
ggplotè il seguente e mostra l'importo medio della tariffa dei viaggi in base all'ora del viaggio: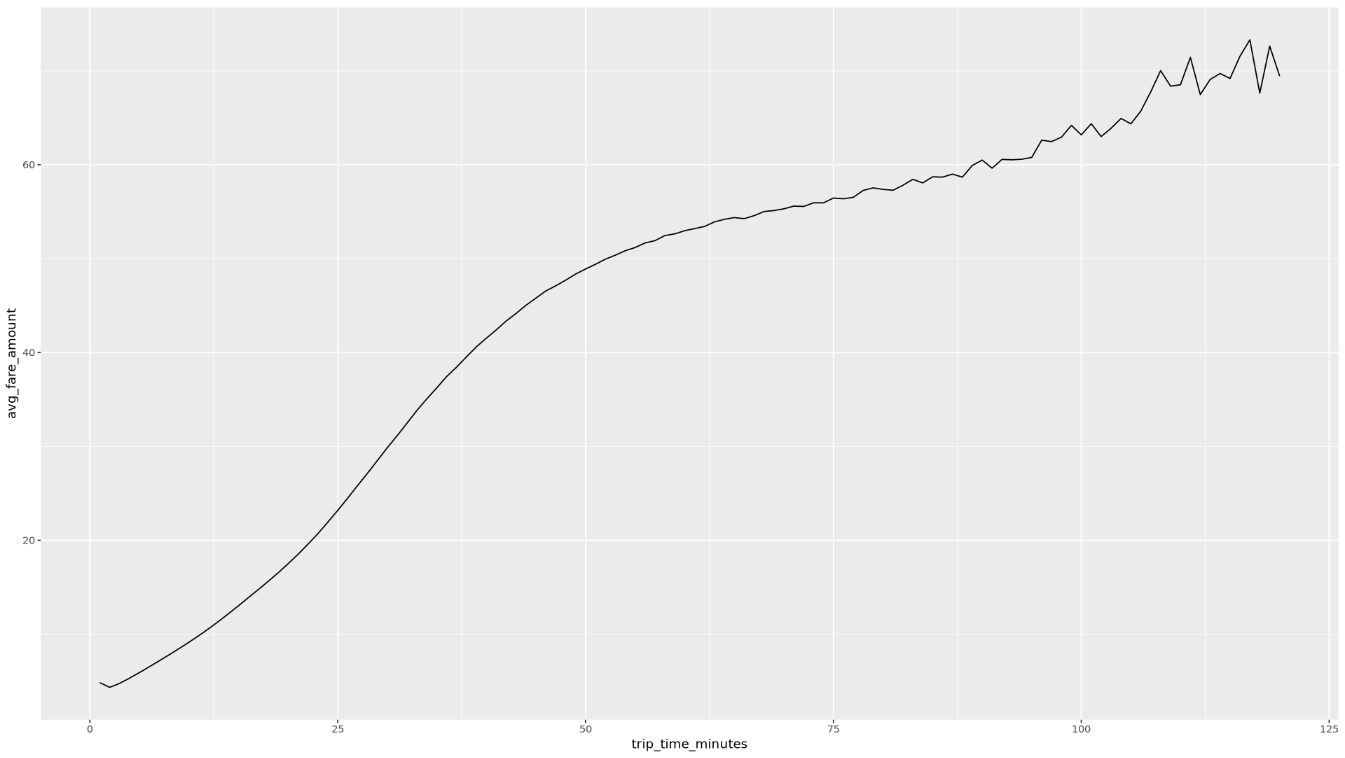
Per altri esempi di visualizzazione con altri campi nei dati, consulta il notebook.
Salvare i dati come file CSV in Cloud Storage
Il passaggio successivo consiste nel salvare i dati estratti da BigQuery come file CSV in Cloud Storage in modo da poterli utilizzare per ulteriori attività di ML.
Nel notebook, carica i dati di addestramento e valutazione da BigQuery in R:
# Prepare training and evaluation data from BigQuery sample_size <- 10000 sql_query <- sprintf(sql_query_template, sample_size) # Split data into 75% training, 25% evaluation train_query <- paste('SELECT * FROM (', sql_query, ') WHERE MOD(CAST(key AS INT64), 100) <= 75') eval_query <- paste('SELECT * FROM (', sql_query, ') WHERE MOD(CAST(key AS INT64), 100) > 75') # Load training data to data frame train_data <- bq_table_download( bq_project_query( PROJECT_ID, query = train_query ) ) # Load evaluation data to data frame eval_data <- bq_table_download( bq_project_query( PROJECT_ID, query = eval_query ) )Controlla il numero di osservazioni in ogni set di dati:
print(paste0("Training instances count: ", nrow(train_data))) print(paste0("Evaluation instances count: ", nrow(eval_data)))Circa il 75% delle istanze totali dovrebbe essere in fase di addestramento, con circa il 25% delle istanze rimanenti in fase di valutazione.
Scrivi i dati in un file CSV locale:
# Write data frames to local CSV files, with headers dir.create(file.path('data'), showWarnings = FALSE) write.table(train_data, "data/train_data.csv", row.names = FALSE, col.names = TRUE, sep = ",") write.table(eval_data, "data/eval_data.csv", row.names = FALSE, col.names = TRUE, sep = ",")Carica i file CSV su Cloud Storage inserendo i comandi
gsutiltra virgolette che vengono passati al sistema:# Upload CSV data to Cloud Storage by passing gsutil commands to system gcs_url <- paste0("gs://", BUCKET_NAME, "/") command <- paste("gsutil mb", gcs_url) system(command) gcs_data_dir <- paste0("gs://", BUCKET_NAME, "/data") command <- paste("gsutil cp data/*_data.csv", gcs_data_dir) system(command) command <- paste("gsutil ls -l", gcs_data_dir) system(command, intern = TRUE)Puoi anche caricare file CSV su Cloud Storage utilizzando la libreria googleCloudStorageR, che richiama l'API JSON di Cloud Storage.
Puoi anche utilizzare bigrquery per scrivere nuovamente i dati da R in BigQuery. La scrittura in BigQuery viene solitamente eseguita dopo aver completato alcune operazioni di preelaborazione o aver generato risultati da utilizzare per ulteriori analisi.
Esegui la pulizia
Per evitare che al tuo account Google Cloud vengano addebitati costi relativi alle risorse utilizzate in questo documento, devi rimuoverle.
Elimina il progetto
Il modo più semplice per eliminare la fatturazione è eliminare il progetto che hai creato. Se intendi esplorare più architetture, tutorial o guide rapide, il riuso dei progetti può aiutarti a non superare i limiti di quota.
- In the Google Cloud console, go to the Manage resources page.
- In the project list, select the project that you want to delete, and then click Delete.
- In the dialog, type the project ID, and then click Shut down to delete the project.
Passaggi successivi
- Scopri di più su come utilizzare i dati di BigQuery nei notebook R nella documentazione di bigrquery.
- Scopri le best practice per l'ingegneria ML in Regole dell'ML.
- Per una panoramica dei principi e dei consigli di architettura specifici per i workload di IA e ML in Google Cloud, consulta la prospettiva IA e ML nel framework di architettura.
- Per altre architetture di riferimento, diagrammi e best practice, visita il Centro architetture di Google Cloud.
Collaboratori
Autore: Alok Pattani | Consulente per gli sviluppatori
Altri collaboratori:
- Jason Davenport | Developer Advocate
- Firat Tekiner | Senior Product Manager

