O uso do executor interativo do Apache Beam com notebooks do JupyterLab permite desenvolver pipelines de maneira iterativa, inspecionar o gráfico do pipeline e analisar PCollections individuais em um fluxo de trabalho de leitura-avaliação-impressão-loop (REPL). Para ver um tutorial que mostra como usar o executor interativo do Apache Beam com notebooks do JupyterLab, consulte Desenvolver com os notebooks do Apache Beam.
Nesta página, você verá detalhes sobre os recursos avançados que podem ser usados com seu notebook do Apache Beam.
FlinkRunner interativo em clusters gerenciados por notebook
Para trabalhar com dados de tamanho de produção interativos do notebook, use o FlinkRunner com algumas opções de pipeline genéricas para instruir a sessão de gerenciamento de clusters duradouros do Dataproc e executar pipelines do Apache Beam de maneira distribuída.
Pré-requisitos
Para usar este recurso, siga estas etapas:
- Ativar a API Dataproc.
- Conceda um papel de administrador ou editor à conta de serviço que executa a instância do notebook para o Dataproc.
- Use um kernel de notebook com a versão 2.40.0 ou posterior do SDK do Apache Beam.
Configuração
Você precisa, no mínimo, das seguintes configurações:
# Set a Cloud Storage bucket to cache source recording and PCollections.
# By default, the cache is on the notebook instance itself, but that does not
# apply to the distributed execution scenario.
ib.options.cache_root = 'gs://<BUCKET_NAME>/flink'
# Define an InteractiveRunner that uses the FlinkRunner under the hood.
interactive_flink_runner = InteractiveRunner(underlying_runner=FlinkRunner())
options = PipelineOptions()
# Instruct the notebook that Google Cloud is used to run the FlinkRunner.
cloud_options = options.view_as(GoogleCloudOptions)
cloud_options.project = 'PROJECT_ID'
Provisionamento explícito (opcional)
Você pode adicionar as seguintes opções.
# Change this if the pipeline needs to run in a different region
# than the default, 'us-central1'. For example, to set it to 'us-west1':
cloud_options.region = 'us-west1'
# Explicitly provision the notebook-managed cluster.
worker_options = options.view_as(WorkerOptions)
# Provision 40 workers to run the pipeline.
worker_options.num_workers=40
# Use the default subnetwork.
worker_options.subnetwork='default'
# Choose the machine type for the workers.
worker_options.machine_type='n1-highmem-8'
# When working with non-official Apache Beam releases, such as Apache Beam built from source
# code, configure the environment to use a compatible released SDK container.
# If needed, build a custom container and use it. For more information, see:
# https://beam.apache.org/documentation/runtime/environments/
options.view_as(PortableOptions).environment_config = 'apache/beam_python3.7_sdk:2.41.0 or LOCATION.pkg.dev/PROJECT_ID/REPOSITORY/your_custom_container'
Uso
# The parallelism is applied to each step, so if your pipeline has 10 steps, you
# end up having 10 * 10 = 100 tasks scheduled, which can be run in parallel.
options.view_as(FlinkRunnerOptions).parallelism = 10
p_word_count = beam.Pipeline(interactive_flink_runner, options=options)
word_counts = (
p_word_count
| 'read' >> ReadWordsFromText('gs://apache-beam-samples/shakespeare/kinglear.txt')
| 'count' >> beam.combiners.Count.PerElement())
# The notebook session automatically starts and manages a cluster to run
# your pipelines with the FlinkRunner.
ib.show(word_counts)
# Interactively adjust the parallelism.
options.view_as(FlinkRunnerOptions).parallelism = 150
# The BigQuery read needs a Cloud Storage bucket as a temporary location.
options.view_as(GoogleCloudOptions).temp_location = ib.options.cache_root
p_bq = beam.Pipeline(runner=interactive_flink_runner, options=options)
delays_by_airline = (
p_bq
| 'Read Dataset from BigQuery' >> beam.io.ReadFromBigQuery(
project=project, use_standard_sql=True,
query=('SELECT airline, arrival_delay '
'FROM `bigquery-samples.airline_ontime_data.flights` '
'WHERE date >= "2010-01-01"'))
| 'Rebalance Data to TM Slots' >> beam.Reshuffle(num_buckets=1000)
| 'Extract Delay Info' >> beam.Map(
lambda e: (e['airline'], e['arrival_delay'] > 0))
| 'Filter Delayed' >> beam.Filter(lambda e: e[1])
| 'Count Delayed Flights Per Airline' >> beam.combiners.Count.PerKey())
# This step reuses the existing cluster.
ib.collect(delays_by_airline)
# Describe the cluster running the pipelines.
# You can access the Flink dashboard from the printed link.
ib.clusters.describe()
# Cleans up all long-lasting clusters managed by the notebook session.
ib.clusters.cleanup(force=True)
Clusters gerenciados pelo notebook
- Por padrão, se você não fornecer opções de pipeline, o Apache Beam interativo sempre reutiliza o cluster usado mais recentemente
para executar um pipeline com o
FlinkRunner.- Para evitar esse comportamento, por exemplo, para executar outro pipeline na mesma
sessão de notebook com um FlinkRunner não hospedado pelo notebook, execute
ib.clusters.set_default_cluster(None).
- Para evitar esse comportamento, por exemplo, para executar outro pipeline na mesma
sessão de notebook com um FlinkRunner não hospedado pelo notebook, execute
- Ao instanciar um novo pipeline que usa um projeto, uma região e uma configuração de provisionamento que mapeia para um cluster atual do Dataproc, o Dataflow também reutiliza o cluster (embora não use o cluster usado mais recentemente).
- No entanto, sempre que uma alteração de provisionamento for realizada, como ao redimensionar um
cluster, ele
será criado para atuar na mudança desejada. Se você pretende redimensionar
um cluster, para evitar o esgotamento dos recursos da nuvem, limpe clusters
desnecessários usando
ib.clusters.cleanup(pipeline). - Quando um Flink
master_urlé especificado, se pertence a um cluster gerenciado pela sessão do notebook, o Dataflow reutiliza o cluster gerenciado.- Se o
master_urlfor desconhecido para a sessão do notebook, isso significa que umFlinkRunnerauto-hospedado pelo usuário é desejado. O notebook não faz nada implicitamente.
- Se o
Solução de problemas
Nesta seção, apresentamos informações para ajudar você a solucionar problemas e depurar o FlinkRunner interativo em clusters gerenciados por notebook.
IOException do Flink: número insuficiente de buffers de rede
Para simplificar, a configuração do buffer de rede do Flink não é exposta para configuração.
Se o gráfico de jobs for muito complicado ou o paralelismo for alto demais, a cardinalidade de etapas multiplicada pelo paralelismo pode ser muito grande, fazer com que muitas tarefas sejam programadas em paralelo e falhar a execução.
Use as dicas a seguir para melhorar a velocidade das execuções interativas:
- Atribua apenas a
PCollectionque você quer inspecionar a uma variável. - Inspecione o recurso
PCollectionsindividualmente. - Use o Reshuffle depois de transformações de alta distribuição de dados
- Ajuste o paralelismo com base no tamanho dos dados (às vezes, menor é mais rápido).
A inspeção dos dados leva muito tempo
Verifique o painel do Flink para o job em execução. Talvez você veja uma etapa em que centenas de tarefas são concluídas, e resta apenas uma porque os dados em trânsito funcionam em uma única máquina e não são embaralhados.
Sempre use a opção "Reshuffle" após uma transformação de alta distribuição de dados, como as seguintes:
- Leitura de linhas de um arquivo
- Leitura de linhas de uma tabela do BigQuery
Sem embaralhamento, os dados de fanout são sempre executados no mesmo worker, e não é possível aproveitar o paralelismo.
De quantos workers eu preciso?
Como regra geral, o cluster do Flink tem o número de vCPUs multiplicado pelo número de slots de worker. Por exemplo, se você tiver 40 workers n1-highmem-8, o cluster do Flink terá no máximo 320 slots, ou 8 será multiplicado por 40.
O ideal é que o worker gerencie um job que leia, mapeie e combine com o paralelismo definido em centenas, o que programa milhares de tarefas em paralelo.
Ele funciona com streaming?
No momento, os pipelines de streaming não são compatíveis com o Flink interativo no recurso de cluster gerenciado pelo notebook.
SQL do Beam e beam_sql mágico
O SQL do Beam permite
consultar PCollections limitadas e ilimitadas com instruções SQL. Se estiver
trabalhando em um notebook do Apache Beam, use o beam_sql
mágico personalizado
do IPython para acelerar o desenvolvimento do pipeline.
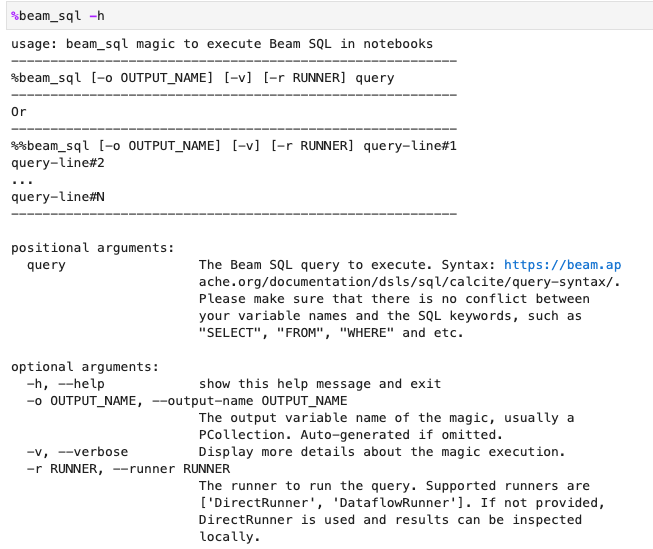
É possível verificar o uso do beam_sql mágico com a opção -h ou --help:
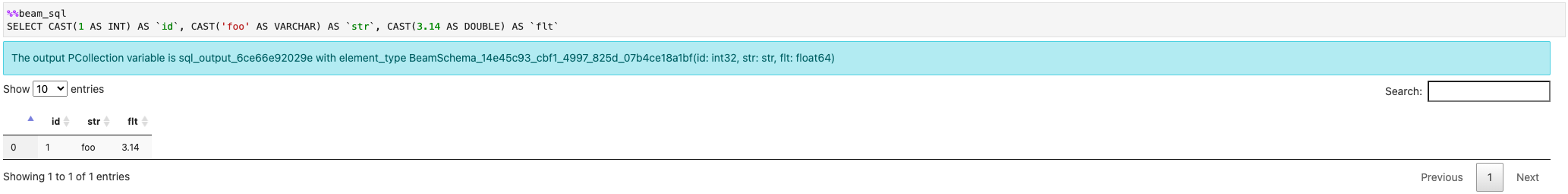
Você pode criar um PCollection a partir de valores constantes:
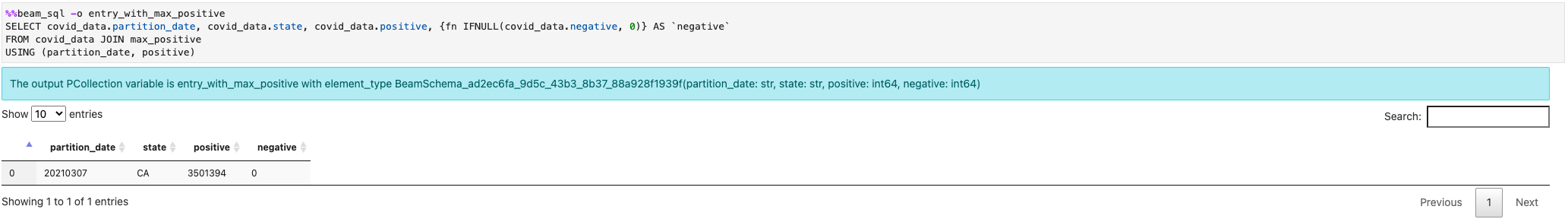
É possível mesclar vários PCollections:
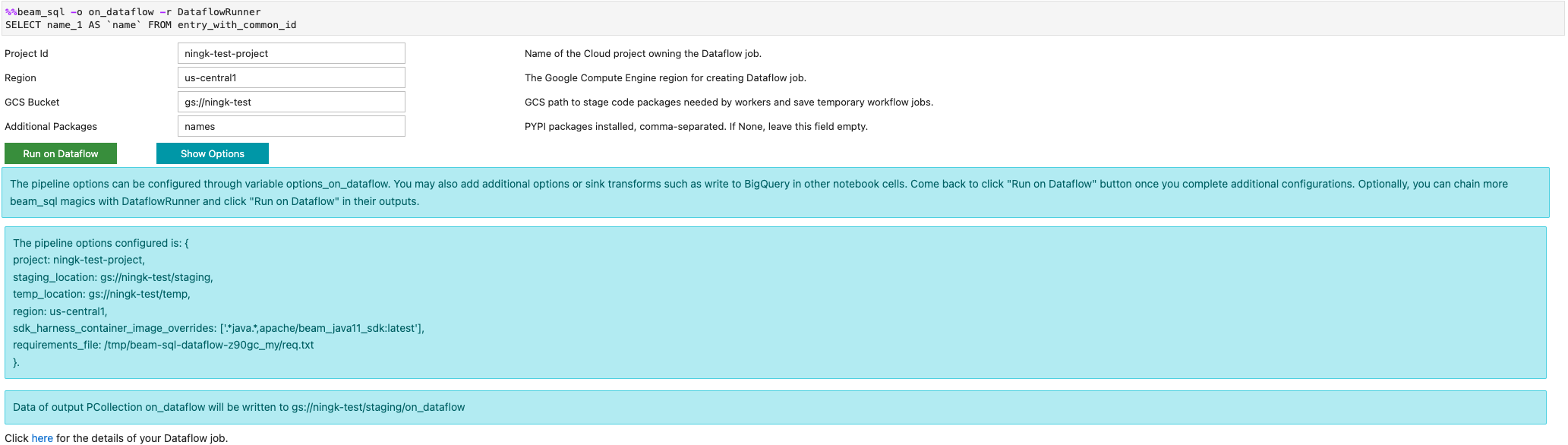
É possível iniciar um job do Dataflow com a opção -r DataflowRunner ou --runner DataflowRunner:
Para saber mais, consulte o notebook de exemplo Apache Beam SQL em notebooks.
Acelerar usando compilador JIT e GPU
É possível usar bibliotecas como numba e
GPUs para acelerar o código do Python e
os pipelines do Apache Beam. Na instância do notebook do Apache Beam criada com
uma GPU nvidia-tesla-t4, para executar em GPUs, compile o código Python com
numba.cuda.jit. Opcionalmente, para acelerar a execução nas CPUs, compile seu
código Python em código de máquina com numba.jit ou numba.njit.
O exemplo a seguir cria um DoFn que é processado em GPUs:
class Sampler(beam.DoFn):
def __init__(self, blocks=80, threads_per_block=64):
# Uses only 1 cuda grid with below config.
self.blocks = blocks
self.threads_per_block = threads_per_block
def setup(self):
import numpy as np
# An array on host as the prototype of arrays on GPU to
# hold accumulated sub count of points in the circle.
self.h_acc = np.zeros(
self.threads_per_block * self.blocks, dtype=np.float32)
def process(self, element: Tuple[int, int]):
from numba import cuda
from numba.cuda.random import create_xoroshiro128p_states
from numba.cuda.random import xoroshiro128p_uniform_float32
@cuda.jit
def gpu_monte_carlo_pi_sampler(rng_states, sub_sample_size, acc):
"""Uses GPU to sample random values and accumulates the sub count
of values within a circle of radius 1.
"""
pos = cuda.grid(1)
if pos < acc.shape[0]:
sub_acc = 0
for i in range(sub_sample_size):
x = xoroshiro128p_uniform_float32(rng_states, pos)
y = xoroshiro128p_uniform_float32(rng_states, pos)
if (x * x + y * y) <= 1.0:
sub_acc += 1
acc[pos] = sub_acc
rng_seed, sample_size = element
d_acc = cuda.to_device(self.h_acc)
sample_size_per_thread = sample_size // self.h_acc.shape[0]
rng_states = create_xoroshiro128p_states(self.h_acc.shape[0], seed=rng_seed)
gpu_monte_carlo_pi_sampler[self.blocks, self.threads_per_block](
rng_states, sample_size_per_thread, d_acc)
yield d_acc.copy_to_host()
A imagem a seguir demonstra o notebook em execução em uma GPU:
Veja mais detalhes no notebook de exemplo Usar GPUs com o Apache Beam.
Criar um contêiner personalizado
Na maioria dos casos, se o pipeline não exigir dependências ou executáveis extras do Python, o Apache Beam poderá usar automaticamente as imagens de contêiner oficiais para executar o código definido pelo usuário. Essas imagens vêm com muitos módulos Python comuns, e não é necessário criá-las ou especificá-las explicitamente.
Em alguns casos, é possível ter dependências extras do Python ou até dependências não Python. Nesses cenários, você pode criar um contêiner personalizado e disponibilizá-lo para o cluster do Flink para execução. Veja na lista a seguir as vantagens de usar um contêiner personalizado:
- Tempo de configuração mais rápido para execuções consecutivas e interativas
- Configurações e dependências estáveis
- Mais flexibilidade: é possível configurar mais dependências do Python
O processo de criação do contêiner pode ser lento, mas é possível fazer tudo no notebook usando o padrão de uso a seguir.
Criar um espaço de trabalho local
Primeiro, crie um diretório de trabalho local no diretório inicial do Jupyter.
!mkdir -p /home/jupyter/.flink
Preparar dependências do Python
Em seguida, instale todas as dependências extras do Python que você pode usar e exporte-as para um arquivo de requisitos.
%pip install dep_a
%pip install dep_b
...
É possível criar explicitamente um arquivo de requisitos usando o comando mágico do notebook %%writefile.
%%writefile /home/jupyter/.flink/requirements.txt
dep_a
dep_b
...
Como alternativa, você pode congelar todas as dependências locais em um arquivo de requisitos. Essa opção pode introduzir dependências não intencionais.
%pip freeze > /home/jupyter/.flink/requirements.txt
Preparar suas dependências não Python
Copie todas as dependências não Python no espaço de trabalho. Se você não tiver dependências não Python, pule esta etapa.
!cp /path/to/your-dep /home/jupyter/.flink/your-dep
...
Criar um DockerFile
Crie um Dockerfile com o comando mágico do notebook %%writefile. Por exemplo:
%%writefile /home/jupyter/.flink/Dockerfile
FROM apache/beam_python3.7_sdk:2.40.0
COPY requirements.txt /tmp/requirements.txt
COPY your_dep /tmp/your_dep
...
RUN python -m pip install -r /tmp/requirements.txt
O contêiner de exemplo usa a imagem do SDK do Apache Beam versão 2.40.0
com o Python 3.7 como base,
adiciona um arquivo your_dep e instala as dependências extras do Python.
Use este Dockerfile como modelo e o edite para seu caso de uso.
Nos pipelines do Apache Beam, ao fazer referência a dependências que não são Python, use os destinos COPY. Por exemplo, /tmp/your_dep é o caminho do arquivo your_dep.
Criar uma imagem de contêiner no Artifact Registry usando o Cloud Build
Ative os serviços do Cloud Build e do Artifact Registry, se ainda não estiverem ativados.
!gcloud services enable cloudbuild.googleapis.com !gcloud services enable artifactregistry.googleapis.comCrie um repositório do Artifact Registry para fazer upload de artefatos. Cada repositório pode conter artefatos para um único formato compatível.
Todo o conteúdo do repositório é criptografado usando chaves de propriedade do Google e gerenciadas pelo Google ou chaves de criptografia gerenciadas pelo cliente. O Artifact Registry usa chaves de propriedade e gerenciadas pelo Google por padrão. Nenhuma configuração é necessária para essa opção.
É preciso ter pelo menos o acesso "Gravador do Artifact Registry" no repositório.
Execute o comando abaixo para criar um novo repositório. O comando usa a sinalização
--asynce retorna imediatamente, sem aguardar a conclusão da operação.gcloud artifacts repositories create REPOSITORY \ --repository-format=docker \ --location=LOCATION \ --asyncSubstitua os seguintes valores:
- REPOSITORY: um nome para o repositório. Para cada local de repositório em um projeto, os nomes dos repositórios precisam ser exclusivos.
- LOCATION: o local do repositório.
Antes de enviar ou extrair imagens, configure o Docker para autenticar solicitações para o Artifact Registry. Para configurar a autenticação nos repositórios do Docker, execute o seguinte comando:
gcloud auth configure-docker LOCATION-docker.pkg.devO comando atualiza a configuração do Docker. Agora é possível se conectar ao Artifact Registry no seu projeto do Google Cloud para enviar imagens.
Use o Cloud Build para criar a imagem do contêiner e salvá-la no Artifact Registry.
!cd /home/jupyter/.flink \ && gcloud builds submit \ --tag LOCATION.pkg.dev/PROJECT_ID/REPOSITORY/flink:latest \ --timeout=20mSubstitua
PROJECT_IDpelo ID do projeto de destino.
Usar contêineres personalizados
Dependendo do executor, é possível usar contêineres personalizados para finalidades diferentes.
Para uso geral do contêiner do Apache Beam, consulte:
Para usar o contêiner do Dataflow, consulte:
Desativar endereços IP externos
Ao criar uma instância de notebook do Apache Beam, para aumentar a segurança, desative endereços IP externos. Como as instâncias de notebook precisam fazer o download de alguns recursos públicos da Internet, como o Artifact Registry, primeiro é necessário criar uma nova rede VPC sem um endereço IP externo. Em seguida, crie um gateway do Cloud NAT para essa rede VPC. Consulte a documentação do Cloud NAT para saber mais. Use a rede VPC e o gateway NAT do Cloud para acessar os recursos de Internet pública necessários sem ativar endereços IP externos.
