L'utilisation de l'exécuteur interactif Apache Beam avec les notebooks JupyterLab vous permet de développer des pipelines de manière itérative, d'inspecter le graphique des pipelines et d'analyser chaque PCollection dans un workflow REPL (read-eval-print-loop). Pour accéder à un tutoriel expliquant comment utiliser l'exécuteur interactif Apache Beam avec les notebooks JupyterLab, consultez la page Développer avec des notebooks Apache Beam.
Cette page fournit des informations sur les fonctionnalités avancées que vous pouvez utiliser avec votre notebook Apache Beam.
Exécuteur FlinkRunner interactif sur les clusters gérés par des notebooks
Pour utiliser des données de production de manière interactive à partir du notebook, vous pouvez utiliser FlinkRunner avec des options de pipeline génériques pour indiquer à la session de notebook de gérer un cluster Dataproc à long terme et d'exécuter vos pipelines Apache Beam de manière distribuée.
Prérequis
Pour utiliser cette fonction :
- activer l'API Dataproc ;
- Attribuez un rôle d'administrateur ou d'éditeur au compte de service qui exécute l'instance de notebook pour Dataproc.
- Utilisez un noyau de notebook avec le SDK Apache Beam version 2.40.0 ou ultérieure.
Configuration
Vous devez au minimum disposer de la configuration suivante:
# Set a Cloud Storage bucket to cache source recording and PCollections.
# By default, the cache is on the notebook instance itself, but that does not
# apply to the distributed execution scenario.
ib.options.cache_root = 'gs://<BUCKET_NAME>/flink'
# Define an InteractiveRunner that uses the FlinkRunner under the hood.
interactive_flink_runner = InteractiveRunner(underlying_runner=FlinkRunner())
options = PipelineOptions()
# Instruct the notebook that Google Cloud is used to run the FlinkRunner.
cloud_options = options.view_as(GoogleCloudOptions)
cloud_options.project = 'PROJECT_ID'
Provisionnement explicite (facultatif)
Vous pouvez ajouter les options suivantes.
# Change this if the pipeline needs to run in a different region
# than the default, 'us-central1'. For example, to set it to 'us-west1':
cloud_options.region = 'us-west1'
# Explicitly provision the notebook-managed cluster.
worker_options = options.view_as(WorkerOptions)
# Provision 40 workers to run the pipeline.
worker_options.num_workers=40
# Use the default subnetwork.
worker_options.subnetwork='default'
# Choose the machine type for the workers.
worker_options.machine_type='n1-highmem-8'
# When working with non-official Apache Beam releases, such as Apache Beam built from source
# code, configure the environment to use a compatible released SDK container.
# If needed, build a custom container and use it. For more information, see:
# https://beam.apache.org/documentation/runtime/environments/
options.view_as(PortableOptions).environment_config = 'apache/beam_python3.7_sdk:2.41.0 or LOCATION.pkg.dev/PROJECT_ID/REPOSITORY/your_custom_container'
Utilisation
# The parallelism is applied to each step, so if your pipeline has 10 steps, you
# end up having 10 * 10 = 100 tasks scheduled, which can be run in parallel.
options.view_as(FlinkRunnerOptions).parallelism = 10
p_word_count = beam.Pipeline(interactive_flink_runner, options=options)
word_counts = (
p_word_count
| 'read' >> ReadWordsFromText('gs://apache-beam-samples/shakespeare/kinglear.txt')
| 'count' >> beam.combiners.Count.PerElement())
# The notebook session automatically starts and manages a cluster to run
# your pipelines with the FlinkRunner.
ib.show(word_counts)
# Interactively adjust the parallelism.
options.view_as(FlinkRunnerOptions).parallelism = 150
# The BigQuery read needs a Cloud Storage bucket as a temporary location.
options.view_as(GoogleCloudOptions).temp_location = ib.options.cache_root
p_bq = beam.Pipeline(runner=interactive_flink_runner, options=options)
delays_by_airline = (
p_bq
| 'Read Dataset from BigQuery' >> beam.io.ReadFromBigQuery(
project=project, use_standard_sql=True,
query=('SELECT airline, arrival_delay '
'FROM `bigquery-samples.airline_ontime_data.flights` '
'WHERE date >= "2010-01-01"'))
| 'Rebalance Data to TM Slots' >> beam.Reshuffle(num_buckets=1000)
| 'Extract Delay Info' >> beam.Map(
lambda e: (e['airline'], e['arrival_delay'] > 0))
| 'Filter Delayed' >> beam.Filter(lambda e: e[1])
| 'Count Delayed Flights Per Airline' >> beam.combiners.Count.PerKey())
# This step reuses the existing cluster.
ib.collect(delays_by_airline)
# Describe the cluster running the pipelines.
# You can access the Flink dashboard from the printed link.
ib.clusters.describe()
# Cleans up all long-lasting clusters managed by the notebook session.
ib.clusters.cleanup(force=True)
Clusters gérés par des notebooks
- Par défaut, si vous ne fournissez aucune option de pipeline, l'interactivité Apache Beam réutilise toujours le cluster utilisé le plus récemment pour exécuter un pipeline avec
FlinkRunner.- Pour éviter ce problème, par exemple pour exécuter un autre pipeline dans la même session de notebook avec un exécuteur FlinkRunner non hébergé par le notebook, exécutez
ib.clusters.set_default_cluster(None).
- Pour éviter ce problème, par exemple pour exécuter un autre pipeline dans la même session de notebook avec un exécuteur FlinkRunner non hébergé par le notebook, exécutez
- Lors de l'instanciation d'un nouveau pipeline utilisant une configuration de projet, de région et de provisionnement mappée à un cluster Dataproc existant, Dataflow réutilise également le cluster (bien qu'il n'utilise pas nécessairement du dernier cluster utilisé).
- Toutefois, chaque fois qu'une modification de provisionnement est appliquée, par exemple lors du redimensionnement d'un cluster, un nouveau cluster est créé pour concrétiser la modification souhaitée. Si vous avez l'intention de redimensionner un cluster, pour éviter d'épuiser les ressources cloud, nettoyez les clusters inutiles à l'aide de
ib.clusters.cleanup(pipeline). - Lorsqu'une URL
master_urlFlink est spécifiée, si elle appartient à un cluster géré par la session de notebook, Dataflow réutilise le cluster géré.- Si
master_urlest inconnue de la session de notebook, cela signifie qu'unFlinkRunnerauto-hébergé par l'utilisateur est souhaité ; le notebook n'effectue aucune action implicite.
- Si
Dépannage
Cette section fournit des informations pour vous aider à résoudre les problèmes et à déboguer l'exécuteur FlinkRunner interactif sur les clusters gérés par des notebooks.
Flink IOException : nombre insuffisant de tampons réseau
Par souci de simplicité, la configuration du tampon réseau Flink n'est pas exposée pour configuration.
Si le graphique de votre tâche est trop compliqué ou que votre parallélisme est trop élevé, la cardinalité des étapes multiplié par le parallélisme peut être trop importante (car trop de tâches sont planifiées en parallèle) et entraîner un échec à l'exécution.
Suivez les conseils ci-dessous pour améliorer la vitesse des exécutions interactives:
- Attribuez uniquement la
PCollectionà inspecter à une variable ; - Inspectez les
PCollectionsune par une. - Utilisez le brassage après des transformations à sortance élevée ;
- Ajustez le parallélisme en fonction de la taille des données (parfois, des données plus petites permettent d'atteindre une vitesse plus élevée).
L'inspection des données prend trop de temps
Consultez la tâche en cours d'exécution dans le tableau de bord Flink. Il se peut que des centaines de tâches soient terminées et qu'une seule reste, car les données en cours de transfert résident sur une seule machine et ne sont pas brassées.
Utilisez toujours le remaniement après une transformation à sortance élevée telle que :
- Lire les lignes d'un fichier
- Lire les lignes d'une table BigQuery
Sans rebrassage, les données de sortance sont toujours exécutées sur le même nœud de calcul et vous ne pouvez pas tirer parti du parallélisme.
De combien de nœuds de calcul ai-je besoin ?
En règle générale, le cluster Flink correspond au nombre de processeurs virtuels multiplié par le nombre d'emplacements de nœuds de calcul. Par exemple, si vous disposez de 40 nœuds de calcul n1-highmem-8, le cluster Flink peut comporter au maximum 320 emplacements, ou 8 multiplié par 40.
Dans l'idéal, le nœud de calcul peut gérer une tâche qui lit, mappe et combine le parallélisme défini dans les centaines, ce qui planifie des milliers de tâches en parallèle.
Fonctionne-t-il avec les flux ?
Les pipelines de traitement par flux ne sont actuellement pas compatibles avec la fonctionnalité interactive Flink sur les clusters gérés par des notebooks.
Beam SQL et magique beam_sql
Beam SQL vous permet d'interroger des PCollectionslimitées et illimitées avec des instructions SQL. Si vous utilisez un notebook Apache Beam, vous pouvez utiliser la commande magique personnalisée IPython beam_sql pour accélérer le développement de votre pipeline.
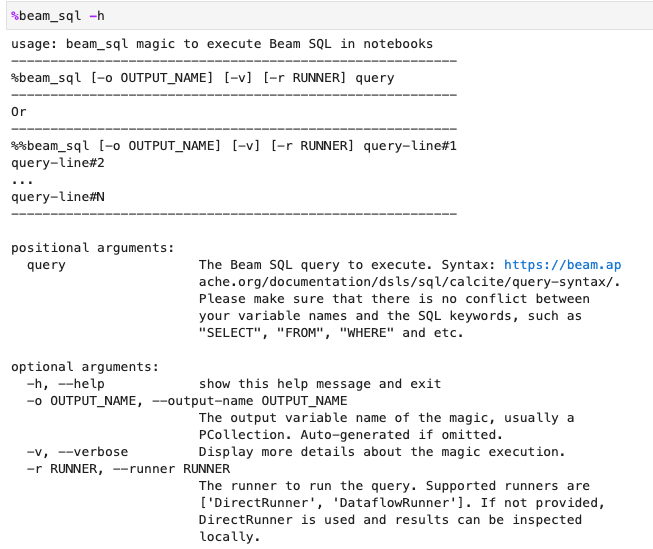
Vous pouvez vérifier l'utilisation de la magique beam_sql avec l'option -h ou --help :
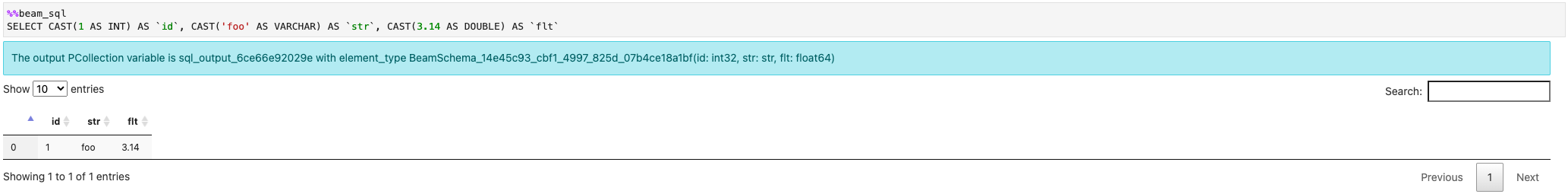
Vous pouvez créer une PCollection à partir de valeurs constantes:
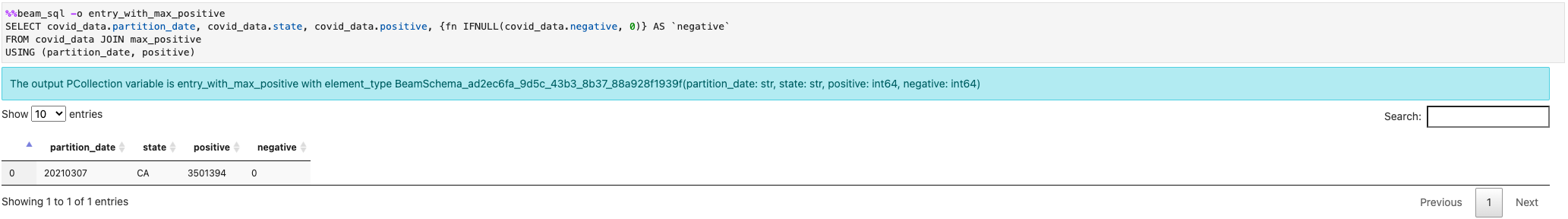
Vous pouvez associer plusieurs PCollections:
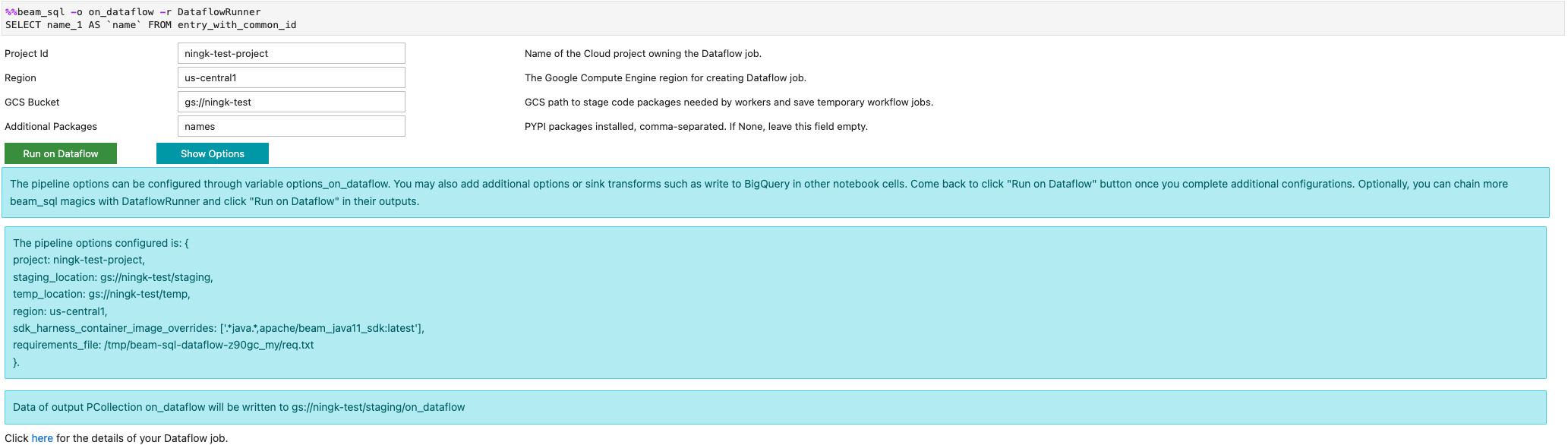
Vous pouvez lancer une tâche Dataflow à l'aide de l'option -r DataflowRunner ou --runner DataflowRunner :
Pour en savoir plus, consultez l'exemple de notebook Apache Beam SQL dans les notebooks.
Accélérer à l'aide d'un compilateur JIT et d'un GPU
Vous pouvez utiliser des bibliothèques telles que numba et des GPU pour accélérer votre code Python et vos pipelines Apache Beam. Dans l'instance de notebook Apache Beam créée avec un GPU nvidia-tesla-t4, compilez votre code Python avec numba.cuda.jit pour exécuter les GPU. Vous pouvez éventuellement compiler le code Python en code machine avec numba.jit ou numba.njit pour accélérer l'exécution.
L'exemple suivant crée un DoFn qui se traite sur les GPU:
class Sampler(beam.DoFn):
def __init__(self, blocks=80, threads_per_block=64):
# Uses only 1 cuda grid with below config.
self.blocks = blocks
self.threads_per_block = threads_per_block
def setup(self):
import numpy as np
# An array on host as the prototype of arrays on GPU to
# hold accumulated sub count of points in the circle.
self.h_acc = np.zeros(
self.threads_per_block * self.blocks, dtype=np.float32)
def process(self, element: Tuple[int, int]):
from numba import cuda
from numba.cuda.random import create_xoroshiro128p_states
from numba.cuda.random import xoroshiro128p_uniform_float32
@cuda.jit
def gpu_monte_carlo_pi_sampler(rng_states, sub_sample_size, acc):
"""Uses GPU to sample random values and accumulates the sub count
of values within a circle of radius 1.
"""
pos = cuda.grid(1)
if pos < acc.shape[0]:
sub_acc = 0
for i in range(sub_sample_size):
x = xoroshiro128p_uniform_float32(rng_states, pos)
y = xoroshiro128p_uniform_float32(rng_states, pos)
if (x * x + y * y) <= 1.0:
sub_acc += 1
acc[pos] = sub_acc
rng_seed, sample_size = element
d_acc = cuda.to_device(self.h_acc)
sample_size_per_thread = sample_size // self.h_acc.shape[0]
rng_states = create_xoroshiro128p_states(self.h_acc.shape[0], seed=rng_seed)
gpu_monte_carlo_pi_sampler[self.blocks, self.threads_per_block](
rng_states, sample_size_per_thread, d_acc)
yield d_acc.copy_to_host()
L'image suivante illustre le notebook exécuté sur un GPU:
Pour en savoir plus, consultez l'exemple de notebook Utiliser des GPU avec Apache Beam.
Créer un conteneur personnalisé
Dans la plupart des cas, si votre pipeline ne nécessite pas de dépendances ou d'exécutables Python supplémentaires, Apache Beam peut automatiquement utiliser ses images de conteneur officielles pour exécuter votre code défini par l'utilisateur. Ces images sont fournies avec de nombreux modules Python courants, sans qu'il soit nécessaire de les créer ou de les spécifier explicitement.
Dans certains cas, vous pouvez avoir des dépendances Python supplémentaires, voire des dépendances non-Python. Dans ces scénarios, vous pouvez créer un conteneur personnalisé et le mettre à la disposition du cluster Flink pour exécution. La liste suivante présente les avantages liés à l'utilisation d'un conteneur personnalisé:
- Réduction du temps de configuration lors des exécutions consécutives/interactives
- Configurations et dépendances stables
- Plus de flexibilité : vous pouvez configurer plus que des dépendances Python
Le processus de création du conteneur peut s'avérer fastidieux, mais vous pouvez tout faire dans le notebook à l'aide du modèle d'utilisation suivant.
Créer un espace de travail local
Commencez par créer un répertoire de travail local dans le répertoire d'accueil de Jupyter.
!mkdir -p /home/jupyter/.flink
Préparer les dépendances Python
Ensuite, installez toutes les dépendances Python supplémentaires que vous pouvez utiliser et exportez-les dans un fichier d'exigences.
%pip install dep_a
%pip install dep_b
...
Vous pouvez créer explicitement un fichier d'exigences à l'aide de la commande magique du notebook %%writefile.
%%writefile /home/jupyter/.flink/requirements.txt
dep_a
dep_b
...
Vous pouvez également geler toutes les dépendances locales dans un fichier d'exigences. Cette option peut introduire des dépendances inattendues.
%pip freeze > /home/jupyter/.flink/requirements.txt
Préparer vos dépendances non-Python
Copiez toutes les dépendances non-Python dans l'espace de travail. Si vous ne disposez d'aucune dépendance non-Python, ignorez cette étape.
!cp /path/to/your-dep /home/jupyter/.flink/your-dep
...
Créer un fichier Dockerfile
Créez un fichier Dockerfile avec la commande magique %%writefile du notebook. Exemple :
%%writefile /home/jupyter/.flink/Dockerfile
FROM apache/beam_python3.7_sdk:2.40.0
COPY requirements.txt /tmp/requirements.txt
COPY your_dep /tmp/your_dep
...
RUN python -m pip install -r /tmp/requirements.txt
L'exemple de conteneur utilise l'image de la version 2.40.0 du SDK Apache Beam avec Python 3.7 comme base, ajoute un fichier your_dep et installe les dépendances Python supplémentaires.
Utilisez ce fichier Dockerfile comme modèle et modifiez-le pour votre cas d'utilisation.
Dans vos pipelines Apache Beam, lorsque vous faites référence à des dépendances non-Python, utilisez leurs destinations COPY. Par exemple, /tmp/your_dep est le chemin d'accès du fichier your_dep.
Créer une image de conteneur dans Artifact Registry à l'aide de Cloud Build
Activez les services Cloud Build et Artifact Registry, si ce n'est pas déjà fait.
!gcloud services enable cloudbuild.googleapis.com !gcloud services enable artifactregistry.googleapis.comCréez un dépôt Artifact Registry afin de pouvoir importer des artefacts. Chaque dépôt peut contenir des artefacts pour un seul format compatible.
L'ensemble du contenu du dépôt est chiffré soit à l'aide de clés appartenant à Google et gérées par Google, soit à l'aide de clés de chiffrement gérées par le client. Artifact Registry utilise par défaut les clés appartenant à Google et gérées par Google, et aucune configuration n'est requise pour cette option.
Vous devez au moins disposer d'un accès Rédacteur à Artifact Registry au dépôt.
Exécutez la commande suivante pour créer un dépôt. La commande utilise l'option
--asyncet affiche immédiatement le résultat, sans attendre la fin de l'opération en cours.gcloud artifacts repositories create REPOSITORY \ --repository-format=docker \ --location=LOCATION \ --asyncRemplacez les valeurs suivantes :
- REPOSITORY : nom de votre dépôt. Pour chaque emplacement de dépôt d'un projet, les noms de dépôt doivent être uniques.
- LOCATION : emplacement de votre dépôt.
Avant de pouvoir transférer ou extraire des images, configurez Docker afin d'authentifier les requêtes envoyées à Artifact Registry. Pour configurer l'authentification auprès des dépôts Docker, exécutez la commande suivante :
gcloud auth configure-docker LOCATION-docker.pkg.devLa commande met à jour votre configuration Docker. Vous pouvez désormais vous connecter à Artifact Registry dans votre projet Google Cloud pour transférer des images.
Utilisez Cloud Build pour créer l'image de conteneur et enregistrez-la dans Artifact Registry.
!cd /home/jupyter/.flink \ && gcloud builds submit \ --tag LOCATION.pkg.dev/PROJECT_ID/REPOSITORY/flink:latest \ --timeout=20mRemplacez
PROJECT_IDpar l'ID de votre projet.
Utiliser des conteneurs personnalisés
Selon l'exécuteur, vous pouvez utiliser des conteneurs personnalisés à différentes fins.
Pour en savoir plus sur l'utilisation générale des conteneurs Apache Beam, consultez les pages suivantes :
Pour l'utilisation des conteneurs Dataflow, consultez les pages suivantes :
Désactiver les adresses IP externes
Lors de la création d'une instance de notebook Apache Beam, désactivez les adresses IP externes pour renforcer la sécurité. Étant donné que les instances de notebook doivent télécharger certaines ressources Internet publiques, telles que Artifact Registry, vous devez d'abord créer un réseau VPC sans adresse IP externe. Ensuite, créez une passerelle Cloud NAT pour ce réseau VPC. Pour en savoir plus sur Cloud NAT, consultez la documentation de Cloud NAT. Utilisez le réseau VPC et la passerelle Cloud NAT pour accéder aux ressources Internet publiques nécessaires sans activer d'adresses IP externes.
