Si usas el ejecutor interactivo de Apache Beam con los cuadernos de JupyterLab, podrás desarrollar flujos de procesamiento de forma iterativa, inspeccionar el gráfico de tu flujo de procesamiento y analizar PCollections individuales en un flujo de trabajo de lectura, evaluación, impresión y bucle (REPL). Para ver un tutorial que muestra cómo usar el ejecutor interactivo de Apache Beam con cuadernos de JupyterLab, consulta Desarrollar con cuadernos de Apache Beam.
En esta página se proporciona información sobre las funciones avanzadas que puedes usar con tu cuaderno de Apache Beam.
FlinkRunner interactivo en clústeres gestionados por cuadernos
Para trabajar con datos de tamaño de producción de forma interactiva desde el cuaderno, puedes usar FlinkRunner con algunas opciones de canalización genéricas para indicar a la sesión del cuaderno que gestione un clúster de Dataproc duradero y que ejecute tus canalizaciones de Apache Beam de forma distribuida.
Requisitos previos
Para usar esta función:
- Habilita la API de Dataproc.
- Asigna un rol de administrador o editor a la cuenta de servicio que ejecuta la instancia del cuaderno de Dataproc.
- Usa un kernel de cuaderno con la versión 2.40.0 o una posterior del SDK de Apache Beam.
Configuración
Como mínimo, debes tener la siguiente configuración:
# Set a Cloud Storage bucket to cache source recording and PCollections.
# By default, the cache is on the notebook instance itself, but that does not
# apply to the distributed execution scenario.
ib.options.cache_root = 'gs://<BUCKET_NAME>/flink'
# Define an InteractiveRunner that uses the FlinkRunner under the hood.
interactive_flink_runner = InteractiveRunner(underlying_runner=FlinkRunner())
options = PipelineOptions()
# Instruct the notebook that Google Cloud is used to run the FlinkRunner.
cloud_options = options.view_as(GoogleCloudOptions)
cloud_options.project = 'PROJECT_ID'
Aprovisionamiento explícito (opcional)
Puedes añadir las siguientes opciones.
# Change this if the pipeline needs to run in a different region
# than the default, 'us-central1'. For example, to set it to 'us-west1':
cloud_options.region = 'us-west1'
# Explicitly provision the notebook-managed cluster.
worker_options = options.view_as(WorkerOptions)
# Provision 40 workers to run the pipeline.
worker_options.num_workers=40
# Use the default subnetwork.
worker_options.subnetwork='default'
# Choose the machine type for the workers.
worker_options.machine_type='n1-highmem-8'
# When working with non-official Apache Beam releases, such as Apache Beam built from source
# code, configure the environment to use a compatible released SDK container.
# If needed, build a custom container and use it. For more information, see:
# https://beam.apache.org/documentation/runtime/environments/
options.view_as(PortableOptions).environment_config = 'apache/beam_python3.7_sdk:2.41.0 or LOCATION.pkg.dev/PROJECT_ID/REPOSITORY/your_custom_container'
Uso
# The parallelism is applied to each step, so if your pipeline has 10 steps, you
# end up having 10 * 10 = 100 tasks scheduled, which can be run in parallel.
options.view_as(FlinkRunnerOptions).parallelism = 10
p_word_count = beam.Pipeline(interactive_flink_runner, options=options)
word_counts = (
p_word_count
| 'read' >> ReadWordsFromText('gs://apache-beam-samples/shakespeare/kinglear.txt')
| 'count' >> beam.combiners.Count.PerElement())
# The notebook session automatically starts and manages a cluster to run
# your pipelines with the FlinkRunner.
ib.show(word_counts)
# Interactively adjust the parallelism.
options.view_as(FlinkRunnerOptions).parallelism = 150
# The BigQuery read needs a Cloud Storage bucket as a temporary location.
options.view_as(GoogleCloudOptions).temp_location = ib.options.cache_root
p_bq = beam.Pipeline(runner=interactive_flink_runner, options=options)
delays_by_airline = (
p_bq
| 'Read Dataset from BigQuery' >> beam.io.ReadFromBigQuery(
project=project, use_standard_sql=True,
query=('SELECT airline, arrival_delay '
'FROM `bigquery-samples.airline_ontime_data.flights` '
'WHERE date >= "2010-01-01"'))
| 'Rebalance Data to TM Slots' >> beam.Reshuffle(num_buckets=1000)
| 'Extract Delay Info' >> beam.Map(
lambda e: (e['airline'], e['arrival_delay'] > 0))
| 'Filter Delayed' >> beam.Filter(lambda e: e[1])
| 'Count Delayed Flights Per Airline' >> beam.combiners.Count.PerKey())
# This step reuses the existing cluster.
ib.collect(delays_by_airline)
# Describe the cluster running the pipelines.
# You can access the Flink dashboard from the printed link.
ib.clusters.describe()
# Cleans up all long-lasting clusters managed by the notebook session.
ib.clusters.cleanup(force=True)
Clústeres gestionados por cuadernos
- De forma predeterminada, si no proporcionas ninguna opción de canalización, Interactive
Apache Beam siempre reutiliza el clúster que se haya usado más recientemente
para ejecutar una canalización con
FlinkRunner.- Para evitar este comportamiento, por ejemplo, para ejecutar otra canalización en la misma sesión del cuaderno con un FlinkRunner no alojado en el cuaderno, ejecuta
ib.clusters.set_default_cluster(None).
- Para evitar este comportamiento, por ejemplo, para ejecutar otra canalización en la misma sesión del cuaderno con un FlinkRunner no alojado en el cuaderno, ejecuta
- Cuando se crea una instancia de una nueva canalización que usa un proyecto, una región y una configuración de aprovisionamiento que se corresponden con un clúster de Dataproc, Dataflow también reutiliza el clúster, aunque es posible que no use el clúster que se haya usado más recientemente.
- Sin embargo, cada vez que se produce un cambio en el aprovisionamiento, como cuando se cambia el tamaño de un clúster, se crea un nuevo clúster para llevar a cabo el cambio deseado. Si tienes intención de cambiar el tamaño de un clúster, usa
ib.clusters.cleanup(pipeline)para eliminar los clústeres innecesarios y evitar agotar los recursos en la nube. - Cuando se especifica un Flink
master_url, si pertenece a un clúster gestionado por la sesión del cuaderno, Dataflow reutiliza el clúster gestionado.- Si
master_urles desconocido para la sesión del cuaderno, significa que se quiere unFlinkRunnerautohospedado por el usuario. El cuaderno no hace nada implícitamente.
- Si
Solución de problemas
En esta sección se proporciona información para ayudarte a solucionar problemas y depurar Interactive FlinkRunner en clústeres gestionados por cuadernos.
Flink IOException: número insuficiente de búferes de red
Para simplificar, la configuración del búfer de red de Flink no se expone para la configuración.
Si el gráfico de la tarea es demasiado complicado o el paralelismo es demasiado alto, la cardinalidad de los pasos multiplicada por el paralelismo puede ser demasiado grande, lo que provoca que se programen demasiadas tareas en paralelo y que falle la ejecución.
Sigue estos consejos para mejorar la velocidad de las ejecuciones interactivas:
- Asigna a una variable solo el
PCollectionque quieras inspeccionar. - Inspecciona
PCollectionsuno por uno. - Usa reshuffle después de transformaciones con un alto fanout.
- Ajusta el paralelismo en función del tamaño de los datos. A veces, cuanto más pequeño, más rápido.
Se tarda demasiado en inspeccionar los datos
Consulta el panel de control de Flink para ver el trabajo en ejecución. Puede que veas un paso en el que se han completado cientos de tareas y solo queda una, porque los datos en curso se encuentran en una sola máquina y no se barajan.
Utiliza siempre la función de reordenación después de una transformación de alta dispersión, como cuando:
- Leer filas de un archivo
- Leer filas de una tabla de BigQuery
Sin la reorganización, los datos de distribución siempre se ejecutan en el mismo trabajador y no puedes aprovechar el paralelismo.
¿Cuántos trabajadores necesito?
Por lo general, el clúster de Flink tiene aproximadamente el número de vCPUs multiplicado por el número de ranuras de trabajador. Por ejemplo, si tienes 40 trabajadores n1-highmem-8, el clúster de Flink tendrá como máximo 320 ranuras (8 multiplicado por 40).
Lo ideal es que el trabajador pueda gestionar una tarea que lea, asigne y combine con un paralelismo de cientos, lo que programa miles de tareas en paralelo.
¿Funciona con streaming?
Actualmente, las canalizaciones de streaming no son compatibles con la función interactiva de Flink en clústeres gestionados por cuadernos.
Beam SQL y beam_sql mágico
Beam SQL te permite consultar PCollections acotados y no acotados con instrucciones SQL. Si estás trabajando en un cuaderno de Apache Beam, puedes usar el comando mágico personalizado de IPython
beam_sql para acelerar el desarrollo de tu canalización.
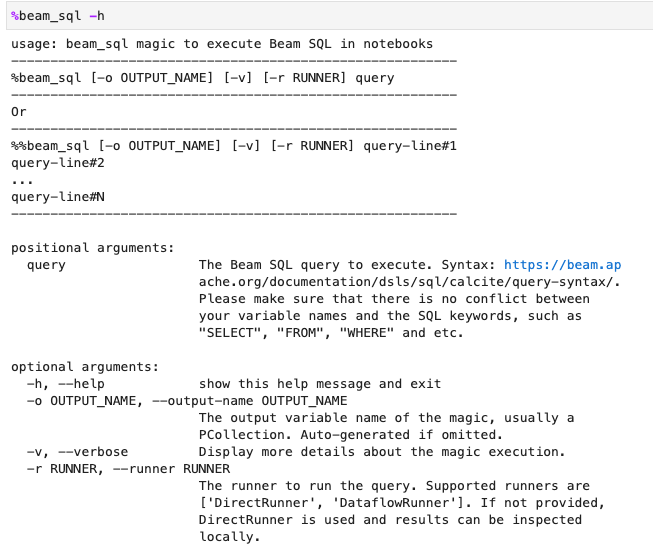
Puedes consultar el uso de la beam_sql magia con la opción -h o --help:
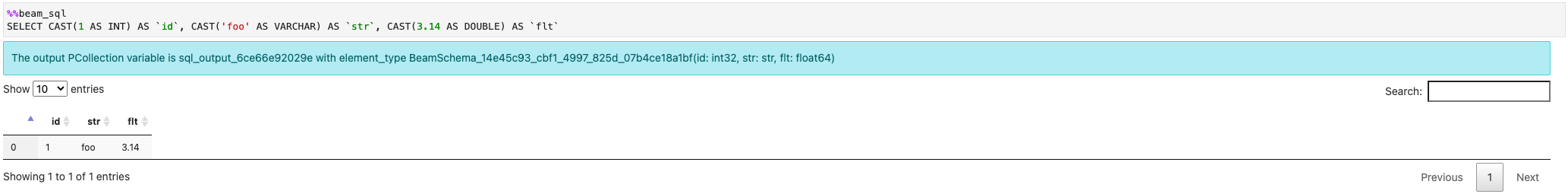
Puedes crear un PCollection a partir de valores constantes:
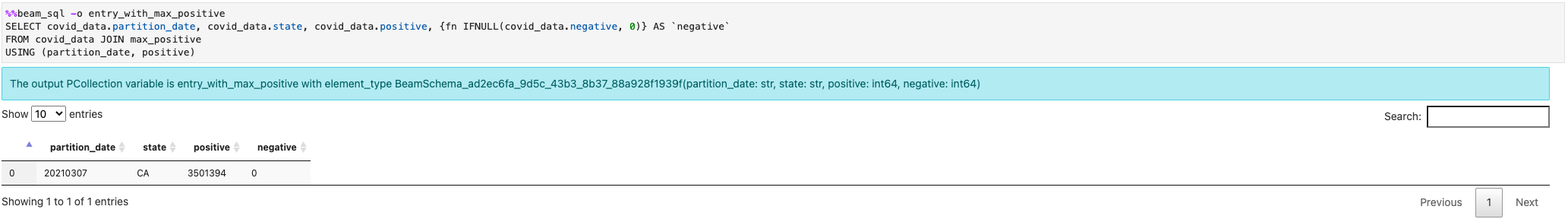
Puedes unirte a varios PCollections:
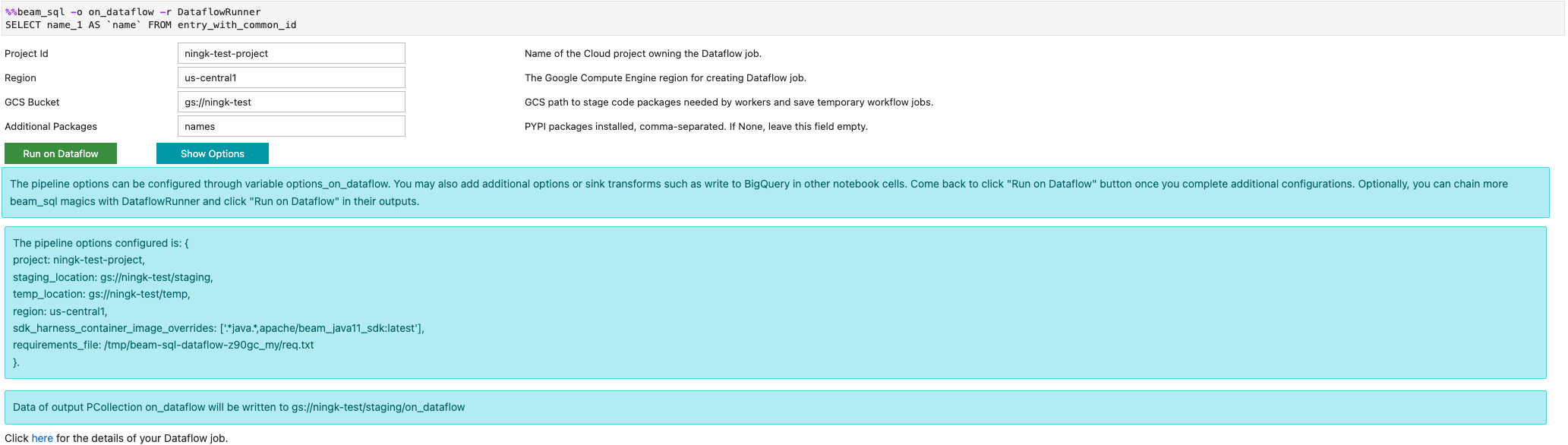
Puedes iniciar una tarea de Dataflow con la opción -r DataflowRunner o --runner DataflowRunner:
Para obtener más información, consulta el cuaderno de ejemplo Apache Beam SQL en cuadernos.
Acelerar el proceso con el compilador JIT y la GPU
Puedes usar bibliotecas como numba y GPUs para acelerar tu código de Python y tus pipelines de Apache Beam. En la instancia de cuaderno de Apache Beam creada con una nvidia-tesla-t4 GPU, para ejecutar el código en GPUs, compila el código de Python con numba.cuda.jit. Opcionalmente, para acelerar la ejecución en las CPUs, compila tu código de Python en código de máquina con numba.jit o numba.njit.
En el siguiente ejemplo se crea un DoFn que se procesa en GPUs:
class Sampler(beam.DoFn):
def __init__(self, blocks=80, threads_per_block=64):
# Uses only 1 cuda grid with below config.
self.blocks = blocks
self.threads_per_block = threads_per_block
def setup(self):
import numpy as np
# An array on host as the prototype of arrays on GPU to
# hold accumulated sub count of points in the circle.
self.h_acc = np.zeros(
self.threads_per_block * self.blocks, dtype=np.float32)
def process(self, element: Tuple[int, int]):
from numba import cuda
from numba.cuda.random import create_xoroshiro128p_states
from numba.cuda.random import xoroshiro128p_uniform_float32
@cuda.jit
def gpu_monte_carlo_pi_sampler(rng_states, sub_sample_size, acc):
"""Uses GPU to sample random values and accumulates the sub count
of values within a circle of radius 1.
"""
pos = cuda.grid(1)
if pos < acc.shape[0]:
sub_acc = 0
for i in range(sub_sample_size):
x = xoroshiro128p_uniform_float32(rng_states, pos)
y = xoroshiro128p_uniform_float32(rng_states, pos)
if (x * x + y * y) <= 1.0:
sub_acc += 1
acc[pos] = sub_acc
rng_seed, sample_size = element
d_acc = cuda.to_device(self.h_acc)
sample_size_per_thread = sample_size // self.h_acc.shape[0]
rng_states = create_xoroshiro128p_states(self.h_acc.shape[0], seed=rng_seed)
gpu_monte_carlo_pi_sampler[self.blocks, self.threads_per_block](
rng_states, sample_size_per_thread, d_acc)
yield d_acc.copy_to_host()
En la siguiente imagen se muestra el cuaderno ejecutándose en una GPU:
Puedes consultar más detalles en el cuaderno de ejemplo Usar GPUs con Apache Beam.
Crear un contenedor personalizado
En la mayoría de los casos, si tu canalización no requiere dependencias ni ejecutables de Python adicionales, Apache Beam puede usar automáticamente sus imágenes de contenedor oficiales para ejecutar el código definido por el usuario. Estas imágenes incluyen muchos módulos de Python comunes, por lo que no tienes que crearlos ni especificarlos explícitamente.
En algunos casos, es posible que tengas dependencias de Python adicionales o incluso dependencias que no sean de Python. En estos casos, puedes crear un contenedor personalizado y ponerlo a disposición del clúster de Flink para que se ejecute. En la siguiente lista se indican las ventajas de usar un contenedor personalizado:
- Tiempo de configuración más rápido para ejecuciones consecutivas e interactivas
- Configuraciones y dependencias estables
- Más flexibilidad: puedes configurar más que dependencias de Python
El proceso de compilación de contenedores puede ser tedioso, pero puedes hacerlo todo en el cuaderno siguiendo el siguiente patrón de uso.
Crear un espacio de trabajo local
Primero, crea un directorio de trabajo local en el directorio principal de Jupyter.
!mkdir -p /home/jupyter/.flink
Preparar dependencias de Python
A continuación, instala todas las dependencias de Python adicionales que puedas usar y expórtalas a un archivo de requisitos.
%pip install dep_a
%pip install dep_b
...
Puedes crear explícitamente un archivo de requisitos mediante el %%writefilecomando mágico de cuaderno.
%%writefile /home/jupyter/.flink/requirements.txt
dep_a
dep_b
...
También puedes congelar todas las dependencias locales en un archivo de requisitos. Esta opción puede introducir dependencias no deseadas.
%pip freeze > /home/jupyter/.flink/requirements.txt
Preparar las dependencias que no son de Python
Copia todas las dependencias que no sean de Python en el espacio de trabajo. Si no tienes ninguna dependencia que no sea de Python, sáltate este paso.
!cp /path/to/your-dep /home/jupyter/.flink/your-dep
...
Crear un Dockerfile
Crea un Dockerfile con el comando mágico %%writefile del cuaderno. Por ejemplo:
%%writefile /home/jupyter/.flink/Dockerfile
FROM apache/beam_python3.7_sdk:2.40.0
COPY requirements.txt /tmp/requirements.txt
COPY your_dep /tmp/your_dep
...
RUN python -m pip install -r /tmp/requirements.txt
El contenedor de ejemplo usa la imagen de la versión 2.40.0 del SDK de Apache Beam con Python 3.7 como base, añade un archivo your_dep e instala las dependencias adicionales de Python.
Usa este Dockerfile como plantilla y edítalo para tu caso práctico.
En tus pipelines de Apache Beam, cuando hagas referencia a dependencias que no sean de Python, usa sus COPY
destinos. Por ejemplo, /tmp/your_dep es la ruta de archivo del archivo your_dep.
Crear una imagen de contenedor en Artifact Registry con Cloud Build
Habilita los servicios Cloud Build y Artifact Registry si aún no lo has hecho.
!gcloud services enable cloudbuild.googleapis.com !gcloud services enable artifactregistry.googleapis.comCrea un repositorio de Artifact Registry para poder subir artefactos. Cada repositorio puede contener artefactos de un solo formato admitido.
Todo el contenido del repositorio se cifra con claves de cifrado gestionadas por el cliente o con Google-owned and Google-managed encryption keys . Artifact Registry usaGoogle-owned and Google-managed encryption keys de forma predeterminada y no es necesario configurar nada para usar esta opción.
Debe tener al menos acceso de escritura de Artifact Registry al repositorio.
Ejecuta el siguiente comando para crear un repositorio. El comando usa la marca
--asyncy se devuelve inmediatamente, sin esperar a que se complete la operación en curso.gcloud artifacts repositories create REPOSITORY \ --repository-format=docker \ --location=LOCATION \ --asyncSustituye los siguientes valores:
- REPOSITORY: un nombre para tu repositorio. Los nombres de los repositorios deben ser únicos en cada ubicación de repositorio de un proyecto.
- LOCATION: la ubicación de tu repositorio.
Antes de enviar o extraer imágenes, configura Docker para autenticar las solicitudes de Artifact Registry. Para configurar la autenticación en repositorios de Docker, ejecuta el siguiente comando:
gcloud auth configure-docker LOCATION-docker.pkg.devEl comando actualiza tu configuración de Docker. Ahora puede conectarse a Artifact Registry en su Google Cloud proyecto para enviar imágenes.
Usa Cloud Build para compilar la imagen de contenedor y guárdala en Artifact Registry.
!cd /home/jupyter/.flink \ && gcloud builds submit \ --tag LOCATION.pkg.dev/PROJECT_ID/REPOSITORY/flink:latest \ --timeout=20mSustituye
PROJECT_IDpor el ID de tu proyecto.
Usar contenedores personalizados
Según el runner, puedes usar contenedores personalizados para diferentes fines.
Para obtener información general sobre el uso de contenedores de Apache Beam, consulta lo siguiente:
Para obtener información sobre el uso de contenedores de Dataflow, consulta lo siguiente:
Inhabilitar direcciones IP externas
Cuando crees una instancia de cuaderno de Apache Beam, inhabilita las direcciones IP externas para aumentar la seguridad. Como las instancias de cuaderno necesitan descargar algunos recursos públicos de Internet, como Artifact Registry, primero debes crear una red de VPC sin una dirección IP externa. A continuación, crea una pasarela de Cloud NAT para esta red de VPC. Para obtener más información sobre Cloud NAT, consulta la documentación de Cloud NAT. Usa la red VPC y la pasarela Cloud NAT para acceder a los recursos de Internet públicos necesarios sin habilitar direcciones IP externas.

