Cette page explique comment exécuter Sentieon® DNASeq® en tant que pipeline Google Cloud pour l'analyse génomique secondaire. Le pipeline correspond aux résultats suivants de la version 3.7 des Bonnes pratiques GATK (Genome Analysis Toolkit) :
- Alignement
- Tri
- Suppression des doublons
- Recalibration du score de qualité des bases (BQSR)
- Appel de variantes
Les formats d'entrée sont les suivants :
- Fichiers fastq
- Fichiers BAM alignés et triés
Objectifs
À la fin de ce tutoriel, vous saurez :
- exécuter un pipeline sur Google Cloud à l'aide de Sentieon® DNAseq® ;
- écrire des fichiers de configuration pour différents cas d'utilisation Sentieon® DNAseq®.
Coûts
Dans ce document, vous utilisez les composants facturables suivants de Google Cloud :
- Compute Engine
- Cloud Storage
Obtenez une estimation des coûts en fonction de votre utilisation prévue à l'aide du simulateur de coût.
Avant de commencer
- Installez Python 2.7 ou une version ultérieure. Pour savoir comment configurer votre environnement de développement Python et installer pip sur votre système, consultez le guide de configuration d'un environnement de développement Python.
- Connectez-vous à votre compte Google Cloud. Si vous débutez sur Google Cloud, créez un compte pour évaluer les performances de nos produits en conditions réelles. Les nouveaux clients bénéficient également de 300 $ de crédits gratuits pour exécuter, tester et déployer des charges de travail.
-
In the Google Cloud console, on the project selector page, select or create a Google Cloud project.
-
Vérifiez que la facturation est activée pour votre projet Google Cloud.
-
Enable the Cloud Life Sciences, Compute Engine, and Cloud Storage APIs.
-
In the Google Cloud console, on the project selector page, select or create a Google Cloud project.
-
Vérifiez que la facturation est activée pour votre projet Google Cloud.
-
Enable the Cloud Life Sciences, Compute Engine, and Cloud Storage APIs.
- Install the Google Cloud CLI.
-
To initialize the gcloud CLI, run the following command:
gcloud init
-
Update and install
gcloudcomponents:gcloud components update
gcloud components install beta - Installez git pour télécharger les fichiers requis.
-
Par défaut, Compute Engine a mis en place des quotas de ressources qui permettent d'éviter toute utilisation accidentelle. En augmentant les quotas, vous pouvez lancer plus de machines virtuelles simultanément, ce qui augmente le débit et réduit les délais d'exécution.
Pour réussir au mieux ce tutoriel, vous devez demander un quota supplémentaire par rapport au quota par défaut de votre projet. La liste suivante contient des recommandations sur l’augmentation des quotas, ainsi que sur les quotas minimaux nécessaires à l’exécution du tutoriel. Effectuez vos demandes de quota dans la région
us-central1:- Processeurs : 64
- Disque persistant standard (Go) : 375
Vous pouvez laisser vides les autres champs de la demande pour conserver vos quotas actuels.
Licence d'évaluation Sentieon®
Avec ce pipeline, Sentieon® vous accorde automatiquement une licence d'évaluation gratuite de deux semaines pour son logiciel, à utiliser avec Google Cloud. Pour recevoir la licence, saisissez votre adresse e-mail dans le champ EMAIL lors de la configuration du pipeline. Pour en savoir plus sur la définition de ce champ, consultez la section Comprendre le format d'entrée.
Pour continuer à utiliser Sentieon® après l'expiration de la licence d'évaluation, veuillez contacter support@sentieon.com.
Configurer votre environnement local et installer les prérequis
Si vous ne disposez pas encore de virtualenv, exécutez la commande suivante pour l'installer à l'aide de pip :
pip install virtualenv
Exécutez la commande suivante pour créer un environnement Python isolé et installer des dépendances :
virtualenv env source env/bin/activate pip install --upgrade \ pyyaml \ google-api-python-client \ google-auth \ google-cloud-storage \ google-auth-httplib2
Télécharger le script de pipeline
Exécutez la commande suivante pour télécharger les exemples de fichiers et définir votre répertoire actuel :
git clone https://github.com/sentieon/sentieon-google-genomics.git cd sentieon-google-genomics
Comprendre le format d'entrée
Le pipeline utilise les paramètres spécifiés dans un fichier JSON en tant qu'entrée.
Le dépôt que vous avez téléchargé comprend un fichier examples/example.json avec le contenu suivant :
{ "FQ1": "gs://sentieon-test/pipeline_test/inputs/test1_1.fastq.gz", "FQ2": "gs://sentieon-test/pipeline_test/inputs/test1_2.fastq.gz", "REF": "gs://sentieon-test/pipeline_test/reference/hs37d5.fa", "OUTPUT_BUCKET": "gs://BUCKET", "ZONES": "us-central1-a,us-central1-b,us-central1-c,us-central1-f", "PROJECT_ID": "PROJECT_ID" "REQUESTER_PROJECT": "PROJECT_ID", "EMAIL": "YOUR_EMAIL_HERE" }
Le tableau suivant décrit les clés JSON dans le fichier :
| Clé JSON | Description |
|---|---|
FQ1 |
La première paire de lectures dans le fichier fastq d'entrée. |
FQ2 |
La deuxième paire de lectures dans le fichier fastq d'entrée. |
BAM |
Le fichier BAM d'entrée, le cas échéant. |
REF |
Le génome de référence. Si définis, les fichiers d’index fastq/BAM sont considérés comme existants. |
OUTPUT_BUCKET |
Le bucket et le répertoire permettant de stocker les données de sortie du pipeline. |
ZONES |
Une liste de zones Google Cloud séparées par une virgule et disponibles pour le nœud de travail. |
PROJECT_ID |
L'ID de votre projet Google Cloud. |
REQUESTER_PROJECT |
Un projet à facturer lors du transfert de données à partir des buckets "Paiements" du demandeur. |
EMAIL |
Votre adresse e-mail |
Exécuter le pipeline
Dans le répertoire
sentieon-google-genomics, modifiez le fichierexamples/example.jsonen remplaçant les variables BUCKET, REQUESTER_PROJECT, EMAIL et PROJECT_ID par les ressources appropriées de votre projet Google Cloud :{ "FQ1": "gs://sentieon-test/pipeline_test/inputs/test1_1.fastq.gz", "FQ2": "gs://sentieon-test/pipeline_test/inputs/test1_2.fastq.gz", "REF": "gs://sentieon-test/pipeline_test/reference/hs37d5.fa", "OUTPUT_BUCKET": "gs://BUCKET", "ZONES": "us-central1-a,us-central1-b,us-central1-c,us-central1-f", "PROJECT_ID": "PROJECT_ID", "REQUESTER_PROJECT": "PROJECT_ID", "EMAIL": "EMAIL_ADDRESS" }
Définissez la variable PROJECT_ID dans votre environnement :
export PROJECT_ID=PROJECT_ID
Saisissez la commande suivante pour exécuter le pipeline DNAseq® sur un petit ensemble de données de test, identifié par les entrées du fichier de configuration. Par défaut, le script vérifie que les fichiers d'entrée existent dans le bucket Cloud Storage avant de démarrer le pipeline.
python runner/sentieon_runner.py --requester_project $PROJECT_ID examples/example.json
Si vous avez spécifié plusieurs essais préemptifs, le pipeline redémarre dès que ses instances sont préemptées. Une fois le pipeline terminé, il envoie un message à la console, indiquant si l'opération a réussi ou échoué.
Configuration recommandée
Dans la plupart des situations, vous pouvez optimiser les délais d'exécution et les coûts en utilisant la configuration suivante. La configuration exécute un séquençage de génome humain 30X pour un coût approximatif de 1,25 $. L'opération prend environ 2 heures. Un séquençage d'exome humain complet coûte environ 0,35 $ et prend 45 minutes environ. Ces deux estimations sont basées sur le fait que les instances du pipeline ne sont pas préemptées.
{ "FQ1": "gs://my-bucket/sample1_1.fastq.gz", "FQ2": "gs://my-bucket/sample1_2.fastq.gz", "REF": "gs://sentieon-test/pipeline_test/reference/hs37d5.fa", "OUTPUT_BUCKET": "gs://BUCKET", "BQSR_SITES": "gs://sentieon-test/pipeline_test/reference/Mills_and_1000G_gold_standard.indels.b37.vcf.gz,gs://sentieon-test/pipeline_test/reference/1000G_phase1.indels.b37.vcf.gz,gs://sentieon-test/pipeline_test/reference/dbsnp_138.b37.vcf.gz", "DBSNP": "gs://sentieon-test/pipeline_test/reference/dbsnp_138.b37.vcf.gz", "PREEMPTIBLE_TRIES": "2", "NONPREEMPTIBLE_TRY": true, "STREAM_INPUT": "True", "ZONES": "us-central1-a,us-central1-b,us-central1-c,us-central1-f", "PROJECT_ID": "PROJECT_ID", "EMAIL": "EMAIL_ADDRESS" }
Options supplémentaires
Vous pouvez personnaliser un pipeline à l'aide des options supplémentaires suivantes.
Options de fichier d'entrée
Le pipeline accepte en entrée plusieurs fichiers fastq séparés par une virgule, comme indiqué dans la configuration suivante :
"FQ1": "gs://my-bucket/s1_prep1_1.fastq.gz,gs://my-bucket/s1_prep2_1.fastq.gz",
"FQ2": "gs://my-bucket/s1_prep1_2.fastq.gz,gs://my-bucket/s1_prep2_2.fastq.gz",
Le pipeline accepte en entrée des fichiers BAM utilisant la clé JSON BAM, séparés par une virgule. Les lectures dans les fichiers BAM ne sont pas alignées sur le génome de référence.
Au lieu de cela, elles commencent à l'étape de déduplication des données du pipeline. L'exemple suivant montre une configuration utilisant deux fichiers BAM en entrée :
"BAM": "gs://my-bucket/s1_prep1.bam,gs://my-bucket/s1_prep2.bam"
Configuration de données ou d'ensembles de données volumineux relatifs aux exomes complets
Les paramètres illustrés dans la configuration recommandée sont optimisés pour les échantillons de génome humain complet séquencés avec une couverture moyenne de 30X. Pour les fichiers beaucoup plus ou beaucoup moins volumineux que les ensembles de données relatives aux génomes complets, vous pouvez réduire ou augmenter les ressources disponibles pour l'instance. Pour de meilleurs résultats avec des ensembles de données volumineux, utilisez les paramètres suivants :
{ "FQ1": "gs://sentieon-test/pipeline_test/inputs/test1_1.fastq.gz", "FQ2": "gs://sentieon-test/pipeline_test/inputs/test1_2.fastq.gz", "REF": "gs://sentieon-test/pipeline_test/reference/hs37d5.fa", "OUTPUT_BUCKET": "gs://BUCKET", "ZONES": "us-central1-a,us-central1-b,us-central1-c,us-central1-f", "PROJECT_ID": "PROJECT_ID", "EMAIL": "EMAIL_ADDRESS", "DISK_SIZE": 600, "MACHINE_TYPE": "n1-highcpu-64", "CPU_PLATFORM": "Intel Broadwell" }
Le tableau suivant fournit une description des paramètres utilisés :
| Clé JSON | Description |
|---|---|
DISK_SIZE |
Espace SSD disponible pour le nœud de travail |
MACHINE_TYPE |
Type de machine virtuelle Compute Engine à utiliser. La valeur par défaut est n1-standard-1. |
CPU_PLATFORM |
Plate-forme de processeur à demander. Il doit s'agir d'un nom de plate-forme de processeur Compute Engine valide (tel que "Intel Skylake"). |
Instances préemptives
Vous pouvez utiliser des instances préemptives dans votre pipeline en spécifiant la clé JSON PREEMPTIBLE_TRIES.
Par défaut, l'exécuteur tente également d'exécuter le pipeline avec une instance standard si le quota d'essais préemptifs a été atteint ou si la clé JSON NONPREEMPTIBLE_TRY est définie sur 0. Vous pouvez désactiver ce comportement en définissant la clé NONPREEMPTIBLE_TRY sur false, comme indiqué dans la configuration suivante :
"PREEMPTIBLE_TRIES": 2,
"NONPREEMPTIBLE_TRY": false
Le tableau suivant fournit une description des paramètres utilisés :
| Clé JSON | Description |
|---|---|
PREEMPTIBLE_TRIES |
Nombre d'essais possibles d'exécution du pipeline avec des instances préemptives |
NONPREEMPTIBLE_TRY |
Détermine si le pipeline doit être exécuté avec une instance standard après que le quota d'essais préemptifs a été atteint |
Groupes de lecture
Des groupes de lecture sont ajoutés lorsque les fichiers fastq sont alignés sur un génome de référence avec Sentieon® BWA. Vous pouvez fournir plusieurs groupes de lecture séparés par une virgule.
Le nombre de groupes de lecture doit correspondre au nombre de fichiers fastq d'entrée.
Le groupe de lecture par défaut est @RG\\tID:read-group\\tSM:sample-name\\tPL:ILLUMINA.
Pour modifier le groupe de lecture, définissez la clé READGROUP dans le fichier d'entrée JSON, comme indiqué dans la configuration suivante :
"READGROUP": "@RG\\tID:my-rgid-1\\tSM:my-sm\\tPL:ILLUMINA,@RG\\tID:my-rgid-2\\tSM:my-sm\\tPL:ILLUMINA"
Le tableau suivant fournit une description du paramètre utilisé :
| Clé JSON | Description |
|---|---|
READGROUP |
Un groupe de lecture contenant des métadonnées d'échantillons |
Pour en savoir plus sur les groupes de lecture, consultez la page Groupes de lecture.
Diffuser des entrées à partir de Cloud Storage
Vous pouvez diffuser des fichiers fastq d'entrée à partir de Cloud Storage, ce qui réduit potentiellement le temps total d'exécution du pipeline. Pour diffuser des fichiers fastq d'entrée à partir de Cloud Storage, définissez la clé JSON STREAM_INPUT sur True :
"STREAM_INPUT": "True"
Le tableau suivant fournit une description du paramètre utilisé :
| Clé JSON | Description |
|---|---|
STREAM_INPUT |
Détermine s'il faut diffuser les fichiers fastq d'entrée directement depuis Google Cloud Storage |
Marquage des doublons
Par défaut, le pipeline supprime les lectures en double des fichiers BAM. Vous pouvez modifier ce comportement en définissant la clé JSON DEDUP, comme indiqué dans la configuration suivante :
"DEDUP": "markdup"
Le tableau suivant fournit une description du paramètre utilisé :
| Clé JSON | Description |
|---|---|
DEDUP |
Comportement de marquage des doublons Valeurs correctes :
|
Recalibration du score de qualité des bases (BQSR) et sites connus
La recalibration du score de qualité des bases requiert des sites connus de variation génétique. Le comportement par défaut consiste à ignorer cette étape du pipeline. Toutefois, vous pouvez activer BSQR en fournissant des sites connus avec la clé JSON BQSR_SITES. S'il est fourni, un fichier DBSNP peut être utilisé pour annoter les variantes de sortie lors des appels de variantes.
"BQSR_SITES": "gs://my-bucket/reference/Mills_and_1000G_gold_standard.indels.b37.vcf.gz,gs://my-bucket/reference/1000G_phase1.indels.b37.vcf.gz,gs://my-bucket/reference/dbsnp_138.b37.vcf.gz",
"DBSNP": "gs://sentieon-test/pipeline_test/reference/dbsnp_138.b37.vcf.gz"
Le tableau suivant fournit une description des paramètres utilisés :
| Clé JSON | Description |
|---|---|
BSQR_SITES |
Active BSQR et utilise une liste de fichiers fournis séparés par une virgule en tant que sites connus |
DBSNP |
Un fichier dbSNP utilisé lors des appels de variantes |
Intervalles
Pour certaines applications, telles que le séquençage ciblé ou d'exome complet, seule une partie du génome est intéressante. Dans ce cas, le fait de spécifier un fichier d'intervalles cibles peut accélérer le traitement et limiter les appels de variantes de faible qualité éloignés de la cible. Vous pouvez utiliser des intervalles avec les clés JSON INTERVAL_FILE et INTERVAL.
"INTERVAL_FILE": "gs://my-bucket/capture-targets.bed",
"INTERVAL": "9:80331190-80646365"
Le tableau suivant fournit une description des paramètres utilisés :
| Clé JSON | Description |
|---|---|
INTERVAL_FILE |
Un fichier contenant des intervalles génomiques à traiter |
INTERVAL |
Une chaîne contenant un intervalle génomique à traiter |
Options de sortie
Par défaut, le pipeline génère un fichier BAM prétraité, des métriques de contrôle qualité et des appels de variantes. Vous pouvez désactiver ces sorties à l'aide des clés JSON NO_BAM_OUTPUT, NO_METRICS et NO_HAPLOTYPER. Si l'argument NO_HAPLOTYPER n'est pas fourni ou défini sur NULL, vous pouvez utiliser la clé JSON GVCF_OUTPUT pour générer des appels de variantes au format gVCF plutôt qu'au format VCF.
"NO_BAM_OUTPUT": "true",
"NO_METRICS": "true",
"NO_HAPLOTYPER": "true",
"GVCF_OUTPUT": "true",
Le tableau suivant fournit une description des paramètres utilisés :
| Clé JSON | Description |
|---|---|
NO_BAM_OUTPUT |
Détermine s'il faut sortir un fichier BAM prétraité |
NO_METRICS |
Détermine s'il faut sortir des métriques de fichier |
NO_HAPLOTYPER |
Détermine s'il faut sortir des appels de variantes |
GVCF_OUTPUT |
Détermine s'il faut sortir des appels de variantes au format gVCF |
Versions de Sentieon® DNAseq®
Vous pouvez utiliser n'importe quelle version récente du package logiciel Sentieon® DNAseq® avec l'API Cloud Life Sciences en spécifiant la clé JSON SENTIEON_VERSION, comme indiqué ci-dessous :
"SENTIEON_VERSION": "201808.08"
Les versions suivantes sont valides :
201711.01201711.02201711.03201711.04201711.05201808201808.01201808.03201808.05201808.06201808.07201808.08
Effectuer un nettoyage
Une fois que vous avez terminé le tutoriel, vous pouvez nettoyer les ressources que vous avez créées sur Google Cloud pour qu'elles ne vous soient plus facturées. Dans les sections suivantes, nous allons voir comment supprimer ou désactiver ces ressources.
Supprimer le projet
Le moyen le plus simple d'empêcher la facturation est de supprimer le projet que vous avez utilisé dans le cadre de ce tutoriel.
Pour supprimer le projet :
- Dans la console Google Cloud, accédez à la page "Projets".
- Dans la liste des projets, sélectionnez celui que vous souhaitez supprimer, puis cliquez sur Supprimer le projet.
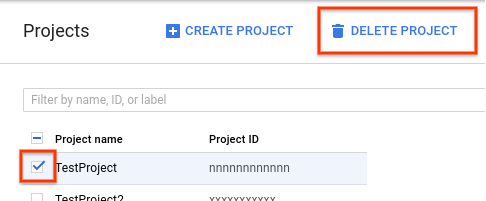
- Dans la boîte de dialogue, saisissez l'ID du projet, puis cliquez sur Arrêter pour supprimer le projet.
Étape suivante
- Pour toute question ou tout problème concernant le pipeline, envoyez un e-mail à l'adresse support@sentieon.com.

