Nesta página, explicamos como executar o Sentieon® DNASeq® como um pipeline do Google Cloud para análise genômica secundária. O pipeline classifica os seguintes resultados com base nas práticas recomendadas do Genome Analysis Toolkit (GATK) versão 3.7:
- Alinhamento
- Classificação
- Remoção duplicada
- Recalibragem do índice de qualidade básico (BQSR, na sigla em inglês)
- Chamada de variantes
Veja abaixo alguns dos formatos de entrada:
- Arquivos fastq
- Arquivos BAM alinhados e classificados
Objetivos
Depois de concluir este tutorial, você saberá:
- como executar um pipeline no Google Cloud usando o Sentieon® DNASeq®;
- como gravar arquivos de configuração para diferentes casos de uso do Sentieon® DNASeq®.
Custos
Neste documento, você usará os seguintes componentes faturáveis do Google Cloud:
- Compute Engine
- Cloud Storage
Para gerar uma estimativa de custo baseada na projeção de uso deste tutorial, use a calculadora de preços.
Antes de começar
- Instale o Python versão 2.7 ou posterior (em inglês). Para mais informações sobre como configurar o ambiente de desenvolvimento Python, como instalar o pip no sistema, consulte o Guia de configuração do ambiente de desenvolvimento Python.
- Faça login na sua conta do Google Cloud. Se você começou a usar o Google Cloud agora, crie uma conta para avaliar o desempenho de nossos produtos em situações reais. Clientes novos também recebem US$ 300 em créditos para executar, testar e implantar cargas de trabalho.
-
No console do Google Cloud, na página do seletor de projetos, selecione ou crie um projeto do Google Cloud.
-
Verifique se a cobrança está ativada para o seu projeto do Google Cloud.
-
Ative as APIs Cloud Life Sciences, Compute Engine, and Cloud Storage.
-
No console do Google Cloud, na página do seletor de projetos, selecione ou crie um projeto do Google Cloud.
-
Verifique se a cobrança está ativada para o seu projeto do Google Cloud.
-
Ative as APIs Cloud Life Sciences, Compute Engine, and Cloud Storage.
- Instale a CLI do Google Cloud.
-
Para inicializar a CLI gcloud, execute o seguinte comando:
gcloud init
- Atualize e instale os componentes
gcloud:gcloud components update
gcloud components install beta - Instale o git para fazer o download dos arquivos necessários.
-
Por padrão, o Compute Engine tem cotas de recursos para impedir o uso inadvertido. Ao aumentar as cotas, você pode iniciar mais máquinas virtuais simultaneamente, aumentando a capacidade e reduzindo o tempo de retorno.
Para os melhores resultados neste tutorial, você precisa solicitar mais cota acima do padrão do seu projeto. Veja recomendações para aumentos de cota na lista a seguir, além das cotas mínimas necessárias para executar o tutorial. Faça as solicitações de cota na região
us-central1:- CPUs: 64
- disco permanente padrão (GB): 375
É possível deixar outros campos de solicitação de cota vazios para manter suas cotas atuais.
Licença de avaliação do Sentieon®
Ao usar esse pipeline, o Sentieon® concede automaticamente uma licença de avaliação gratuita
do software de duas semanas
para usar com o Google Cloud. Para receber a licença, insira seu endereço de e-mail no campo EMAIL ao configurar o pipeline. Consulte Como funciona o formato de entrada para mais informações sobre como definir este campo.
Para continuar usando o Sentieon® depois que a licença de avaliação expirar, entre em contato com support@sentieon.com.
Configurar o ambiente local e instalar pré-requisitos
Se você não tiver o virtualenv, execute o seguinte comando para instalá-lo usando o pip:
pip install virtualenv
Execute este comando para criar um ambiente Python isolado e instalar as dependências:
virtualenv env source env/bin/activate pip install --upgrade \ pyyaml \ google-api-python-client \ google-auth \ google-cloud-storage \ google-auth-httplib2
Como fazer o download do script do canal
Execute o seguinte comando para fazer o download dos arquivos de exemplo e definir seu diretório atual:
git clone https://github.com/sentieon/sentieon-google-genomics.git cd sentieon-google-genomics
Noções básicas sobre o formato de entrada
O canal usa como entrada parâmetros especificados em um arquivo JSON.
No repositório do que você fez o download, há um arquivo examples/example.json com o seguinte conteúdo:
{
"FQ1": "gs://sentieon-test/pipeline_test/inputs/test1_1.fastq.gz",
"FQ2": "gs://sentieon-test/pipeline_test/inputs/test1_2.fastq.gz",
"REF": "gs://sentieon-test/pipeline_test/reference/hs37d5.fa",
"OUTPUT_BUCKET": "gs://BUCKET",
"ZONES": "us-central1-a,us-central1-b,us-central1-c,us-central1-f",
"PROJECT_ID": "PROJECT_ID"
"REQUESTER_PROJECT": "PROJECT_ID",
"EMAIL": "YOUR_EMAIL_HERE"
}
Na tabela a seguir, você verá a descrição das chaves JSON no arquivo:
| Chave JSON | Descrição |
|---|---|
FQ1 |
É o primeiro par de leituras no arquivo fastq de entrada. |
FQ2 |
É o segundo par de leituras no arquivo fastq de entrada. |
BAM |
O arquivo BAM de entrada, se aplicável. |
REF |
O genoma de referência. Se definido, presume-se que existam os arquivos de índice fastq/BAM. |
OUTPUT_BUCKET |
O bucket e o diretório usados para armazenar a saída de dados do canal. |
ZONES |
Uma lista separada por vírgulas de zonas do Google Cloud para usar para o nó de trabalho. |
PROJECT_ID |
É o ID do seu projeto no Google Cloud. |
REQUESTER_PROJECT |
Um projeto a ser faturado ao transferir dados dos buckets de pagamentos do solicitante. |
EMAIL |
Endereço de e-mail. |
Executar o canal
No diretório
sentieon-google-genomics, edite o arquivoexamples/example.jsonsubstituindo as variáveis BUCKET, REQUESTER_PROJECT, EMAIL e PROJECT_ID pelos recursos relevantes do projeto do Google Cloud:{ "FQ1": "gs://sentieon-test/pipeline_test/inputs/test1_1.fastq.gz", "FQ2": "gs://sentieon-test/pipeline_test/inputs/test1_2.fastq.gz", "REF": "gs://sentieon-test/pipeline_test/reference/hs37d5.fa", "OUTPUT_BUCKET": "gs://BUCKET", "ZONES": "us-central1-a,us-central1-b,us-central1-c,us-central1-f", "PROJECT_ID": "PROJECT_ID", "REQUESTER_PROJECT": "PROJECT_ID", "EMAIL": "EMAIL_ADDRESS" }Defina a variável PROJECT_ID no seu ambiente:
export PROJECT_ID=PROJECT_ID
Aplique o comando a seguir para executar a pipeline do DNASeq® em um pequeno conjunto de dados de teste identificado pelas entradas no arquivo de configuração. Por padrão, o script verifica se os arquivos de entrada existem no seu bucket do Cloud Storage antes de iniciar o canal.
python runner/sentieon_runner.py --requester_project $PROJECT_ID examples/example.json
Se você especificou várias tentativas preemptivas, o pipeline é reiniciado sempre que a interrupção das instâncias é forçada. Após o término do canal, ele envia uma mensagem ao console informando se o canal foi bem-sucedido ou não.
Configuração recomendada
Para a maioria das situações, você pode otimizar o tempo de retorno e o custo usando a configuração a seguir. A configuração executa um genoma humano 30 vezes a um custo de aproximadamente US$ 1,25 e leva cerca de duas horas. Um exoma humano inteiro custa aproximadamente US$ 0,35 e leva cerca de 45 minutos. As duas estimativas são baseadas em instâncias do canal não sendo antecipadas.
{
"FQ1": "gs://my-bucket/sample1_1.fastq.gz",
"FQ2": "gs://my-bucket/sample1_2.fastq.gz",
"REF": "gs://sentieon-test/pipeline_test/reference/hs37d5.fa",
"OUTPUT_BUCKET": "gs://BUCKET",
"BQSR_SITES": "gs://sentieon-test/pipeline_test/reference/Mills_and_1000G_gold_standard.indels.b37.vcf.gz,gs://sentieon-test/pipeline_test/reference/1000G_phase1.indels.b37.vcf.gz,gs://sentieon-test/pipeline_test/reference/dbsnp_138.b37.vcf.gz",
"DBSNP": "gs://sentieon-test/pipeline_test/reference/dbsnp_138.b37.vcf.gz",
"PREEMPTIBLE_TRIES": "2",
"NONPREEMPTIBLE_TRY": true,
"STREAM_INPUT": "True",
"ZONES": "us-central1-a,us-central1-b,us-central1-c,us-central1-f",
"PROJECT_ID": "PROJECT_ID",
"EMAIL": "EMAIL_ADDRESS"
}
Outras opções
Você pode personalizar um canal usando as seguintes opções adicionais.
Opções de arquivo de entrada
O pipeline é compatível com vários arquivos fastq separados por vírgula como entrada, conforme mostrado na seguinte configuração:
"FQ1": "gs://my-bucket/s1_prep1_1.fastq.gz,gs://my-bucket/s1_prep2_1.fastq.gz",
"FQ2": "gs://my-bucket/s1_prep1_2.fastq.gz,gs://my-bucket/s1_prep2_2.fastq.gz",
O pipeline aceita arquivos BAM separados por vírgulas como entrada usando a chave
JSON BAM. As leituras nos arquivos BAM não estão alinhadas ao genoma de referência.
Em vez disso, elas começam no estágio de eliminação de duplicação de dados do pipeline. O seguinte
exemplo mostra uma configuração que usa dois arquivos BAM como entrada:
"BAM": "gs://my-bucket/s1_prep1.bam,gs://my-bucket/s1_prep2.bam"
Dados do exoma inteiro ou grandes conjuntos de dados
As configurações da configuração recomendada são otimizadas para amostras de genoma humano completo sequenciadas para uma cobertura média de 30x. Para arquivos muito menores ou maiores que os conjuntos de dados padrão do genoma inteiro, você pode aumentar ou diminuir os recursos disponíveis para a instância. Para ter os melhores resultados com grandes conjuntos de dados, use as configurações a seguir:
{
"FQ1": "gs://sentieon-test/pipeline_test/inputs/test1_1.fastq.gz",
"FQ2": "gs://sentieon-test/pipeline_test/inputs/test1_2.fastq.gz",
"REF": "gs://sentieon-test/pipeline_test/reference/hs37d5.fa",
"OUTPUT_BUCKET": "gs://BUCKET",
"ZONES": "us-central1-a,us-central1-b,us-central1-c,us-central1-f",
"PROJECT_ID": "PROJECT_ID",
"EMAIL": "EMAIL_ADDRESS",
"DISK_SIZE": 600,
"MACHINE_TYPE": "n1-highcpu-64",
"CPU_PLATFORM": "Intel Broadwell"
}
Na tabela a seguir, você verá uma descrição das configurações usadas:
| Chave JSON | Descrição |
|---|---|
DISK_SIZE |
Espaço SSD disponível para o nó de trabalho. |
MACHINE_TYPE |
O tipo de máquina virtual do Compute Engine a ser usado. Padrões para n1-standard-1. |
CPU_PLATFORM |
A plataforma de CPU a ser solicitada. Precisa ser um nome válido de plataforma de CPU do Compute Engine, como o Intel Skylake. |
Instâncias preemptivas
Para usar instâncias preemptivas no pipeline,
configure a chave JSON PREEMPTIBLE_TRIES.
Por padrão, o executor tentará executar o canal com uma instância padrão se as tentativas preemptivas estiverem esgotadas ou se a chave JSON NONPREEMPTIBLE_TRY estiver definida como 0. Para desativar esse comportamento, defina a
chave NONPREEMPTIBLE_TRY como false, conforme mostrado na seguinte configuração:
"PREEMPTIBLE_TRIES": 2,
"NONPREEMPTIBLE_TRY": false
Na tabela a seguir, você verá uma descrição das configurações usadas:
| Chave JSON | Descrição |
|---|---|
PREEMPTIBLE_TRIES |
O número de testes possíveis do canal ao usar instâncias preemptivas. |
NONPREEMPTIBLE_TRY |
Determina se você deve tentar executar o canal com uma instância padrão depois que as tentativas preemptivas estiverem esgotadas. |
Grupos de leitura
Os grupos de leitura são adicionados quando os arquivos fastq são alinhados a um genoma de referência
usando o Sentieon® BWA. É possível fornecer vários grupos de leitura separados por vírgulas.
O número de grupos de leitura precisa corresponder ao número de arquivos fastq de entrada.
O grupo de leitura padrão é @RG\\tID:read-group\\tSM:sample-name\\tPL:ILLUMINA.
Para alterar o grupo de leitura, defina a chave READGROUP no arquivo de entrada JSON, conforme
mostrado na seguinte configuração:
"READGROUP": "@RG\\tID:my-rgid-1\\tSM:my-sm\\tPL:ILLUMINA,@RG\\tID:my-rgid-2\\tSM:my-sm\\tPL:ILLUMINA"
Na tabela a seguir, você verá uma descrição da configuração usada:
| Chave JSON | Descrição |
|---|---|
READGROUP |
Um grupo de leitura com metadados de amostra. |
Para mais informações sobre grupos de leitura, consulte Grupos de leitura.
Entrada de streaming do Cloud Storage
É possível transmitir arquivos fastq de entrada do Cloud Storage, o que pode reduzir o tempo de execução total do pipeline. Para transmitir arquivos fastq de entrada do Cloud Storage, defina a chave STREAM_INPUT JSON como True:
"STREAM_INPUT": "True"
Na tabela a seguir, você verá uma descrição da configuração usada:
| Chave JSON | Descrição |
|---|---|
STREAM_INPUT |
Determina se os arquivos fastq de entrada devem ser transmitidos diretamente do Cloud Storage. |
Marcação duplicada
Por padrão, o canal remove leituras duplicadas de arquivos BAM. É possível
alterar esse comportamento definindo a chave JSON DEDUP, conforme mostrado na seguinte
configuração:
"DEDUP": "markdup"
Na tabela a seguir, você verá uma descrição da configuração usada:
| Chave JSON | Descrição |
|---|---|
DEDUP |
Comportamento de marcação duplicado. Valores válidos:
|
Recalibragem do índice de qualidade básica (BQSR, na sigla em inglês) e sites conhecidos
O BSQR requer sites conhecidos de variação genética. O comportamento padrão é pular essa etapa do pipeline. No entanto, é possível ativar a BSQR fornecendo sites conhecidos com a chave JSON BQSR_SITES. Se fornecido, um arquivo DBSNP pode ser usado para anotar as variantes de saída durante a chamada de variantes.
"BQSR_SITES": "gs://my-bucket/reference/Mills_and_1000G_gold_standard.indels.b37.vcf.gz,gs://my-bucket/reference/1000G_phase1.indels.b37.vcf.gz,gs://my-bucket/reference/dbsnp_138.b37.vcf.gz",
"DBSNP": "gs://sentieon-test/pipeline_test/reference/dbsnp_138.b37.vcf.gz"
Na tabela a seguir, você verá uma descrição das configurações usadas:
| Chave JSON | Descrição |
|---|---|
BSQR_SITES |
Ativa o BSQR e usa uma lista separada por vírgulas de arquivos fornecidos como sites conhecidos. |
DBSNP |
Um arquivo dbSNP usado durante a chamada de variantes. |
Intervalos
Para algumas aplicações, como sequenciamento direcionado ou exoma completo, talvez você queira apenas uma parte do genoma. Nesses casos, fornecer um arquivo de intervalos de destino pode acelerar o processamento e reduzir chamadas de variantes fora da meta e de baixa qualidade. Você pode usar intervalos com as chaves JSON INTERVAL_FILE e INTERVAL.
"INTERVAL_FILE": "gs://my-bucket/capture-targets.bed",
"INTERVAL": "9:80331190-80646365"
Na tabela a seguir, você verá uma descrição das configurações usadas:
| Chave JSON | Descrição |
|---|---|
INTERVAL_FILE |
Um arquivo com intervalos genômicos a serem processados. |
INTERVAL |
Uma string com um intervalo genômico a ser processado. |
Opções de saída
Por padrão, o pipeline produz um BAM pré-processado, métricas de controle de qualidade e chamadas variantes. É possível desativar qualquer uma dessas saídas usando as chaves JSON NO_BAM_OUTPUT, NO_METRICS e NO_HAPLOTYPER. Se o argumento NO_HAPLOTYPER não for fornecido ou for NULL, será possível utilizar a chave JSON GVCF_OUTPUT para produzir chamadas variantes no formato gVCF, em vez de no formato VCF.
"NO_BAM_OUTPUT": "true",
"NO_METRICS": "true",
"NO_HAPLOTYPER": "true",
"GVCF_OUTPUT": "true",
Na tabela a seguir, você verá uma descrição das configurações usadas:
| Chave JSON | Descrição |
|---|---|
NO_BAM_OUTPUT |
Determina se um arquivo BAM pré-processado deve ser enviado. |
NO_METRICS |
Determina se métricas de arquivo devem ser enviadas. |
NO_HAPLOTYPER |
Determina se deve chamadas variantes devem ser enviadas. |
GVCF_OUTPUT |
Determina se chamadas variantes no formato gVCF devem ser enviadas. |
Versões do Sentieon® DNASeq®
É possível usar qualquer versão recente do pacote de software Sentieon® DNASeq® com a
API Cloud Life Sciences especificando a chave JSON SENTIEON_VERSION da seguinte
maneira:
"SENTIEON_VERSION": "201808.08"
Estas versões são válidas:
201711.01201711.02201711.03201711.04201711.05201808201808.01201808.03201808.05201808.06201808.07201808.08
Limpeza
Depois de concluir o tutorial, limpe os recursos criados no Google Cloud para não ser cobrado por eles no futuro. Nas seções a seguir, você aprenderá a excluir e desativar esses recursos.
Exclua o projeto
A maneira mais fácil de evitar a cobrança é excluir o projeto usado no tutorial.
Para excluir o projeto, faça o seguinte:
- No Console do Google Cloud, acesse a página "Projetos".
-
Na lista de projetos, selecione o que você quer excluir e clique em Excluir projeto.
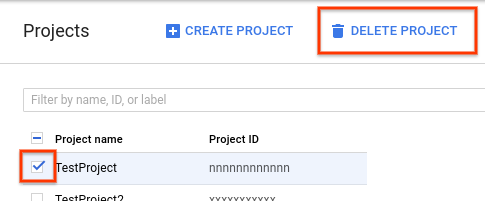
- Na caixa de diálogo, digite o código do projeto e clique em Encerrar para excluí-lo.
A seguir
- Caso tenha dúvidas sobre o canal ou encontre algum problema, envie um e-mail para support@sentieon.com.
