Hail、BigQuery、Dataproc でのゲノム解析
Google Cloud Japan Team
※この投稿は米国時間 2020 年 7 月 2 日に、Google Cloud blog に投稿されたものの抄訳です。
Google Cloud では、大規模な研究プロジェクトを実施している組織を支援しています。Google Cloud が提供するゲノム解析に最適なソリューションにより、研究者は、新しい治療方法、オーダーメイド医療、新薬開発の研究に集中して全力で取り組むことができます(ゲノムデータ解析アーキテクトの作成に関する詳細は、こちらの投稿をご覧ください)。
Hail は、オープンソースかつ汎用の Python ベースのデータ分析ライブラリです。多様なデータタイプとメソッドを備え、Apache Sparks を基盤としてゲノムデータを処理できます。Hail は、多次元構造化データ(ゲノムワイド関連解析(GWAS)のゲノムデータなど)に対応して最上級のサポートを提供することを目的に構築されました。Hail チームがそのソフトウェアを MIT ライセンスによって利用できるようにしたことで、Hail はゲノムデータ処理ツールのスイートである Google Cloud Life Sciences にとっての完璧な拡張機能となりました。Dataproc は、クラウドにおけるオープンソース データ処理や分析処理の高速化、簡素化、セキュリティ強化を実現します。また、独自設計のクラスタによってデータ サイエンスの処理速度を高めるフルマネージド Apache Spark を提供します。
Google Cloud が他のクラウド コンピューティング プラットフォームよりも抜きん出て優れている点は、ゲノムデータと他の医療システムのデータセットを簡単にマージできる医療向けツールにあります。遺伝子型データが電子カルテの表現型データ、デバイスデータ、医療用メモ、医療用画像とハーモナイズされると、仮説空間は無限になります。さらに、AI Platform Notebooks や Dataproc Hub などの Google Cloud ベースの分析プラットフォームでは、研究者が最先端の ML ツール使用して共同作業をスムーズに進めることができるうえ、安全かつ適切な方法でデータセットを結びつけることができます。
Hail と Dataproc のスタートガイド
Hail バージョン 0.2.15 より、Hail の pip インストールにコマンドライン ツール hailctl がバンドルされました。これには Hail 用に構成された Dataproc クラスタで動作する Dataproc というサブモジュールが含まれています。つまり、Google Cloud Console に移動して、コンソール ウィンドウ上部の Cloud Shell のアイコンをクリックするだけで、Dataproc と Hail を開始できます。この Cloud Shell では、ブラウザから直接クラウド リソースにコマンドラインでアクセスでき、ツールをシステムにインストールする必要がありません。このシェルから、以下のように入力して Hail をすぐにインストールできます。Hail をダウンロードしてインストールしたら、次のコマンドを実行するだけで、Apache Spark と Hail 用に構成された Dataproc クラスタを作成できます。
Dataproc クラスタを作成したら、Cloud Shell の [エディタを開く] ボタンをクリックして組み込みのエディタを開き、コードの作成や変更を行うことができます。このエディタで [新しいファイル] を選択して、「my-first-hail-job.py」というファイルを呼び出します。

エディタで、次のコードをコピーして貼り付けます。
このファイルは Cloud Shell エディタによってデフォルトの場所に保存されますが、メニューからファイルの作成場所を指定してファイルを保存することもできます。ファイルが保存されたことを確認したら、[ターミナルを開く] ボタンをクリックしてコマンドライン ターミナルに戻ります。
ターミナルから、次のコマンドを実行します。
ジョブが開始したら、Google Cloud Console の [Dataproc] セクションに移動し、[ジョブ] タブでゲノム解析ジョブの出力を確認します。
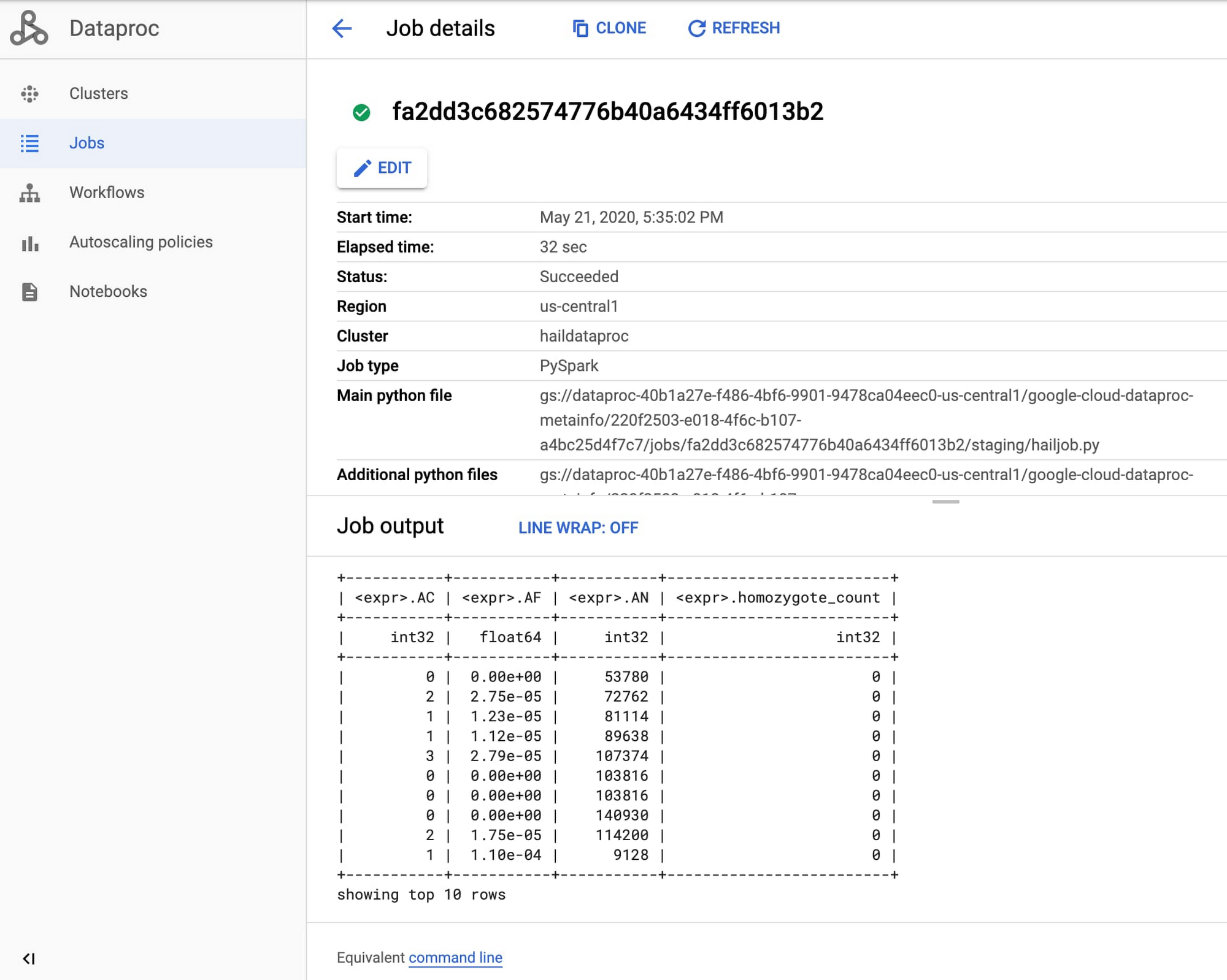

これで、Dataproc 上で最初の Hail ジョブを実行できました。詳しくは、Using Hail on Google Cloud Platform(Google Cloud Platform で Hail を使用する)をご覧ください。
次に、Dataproc と Hail を他の臨床データ ウェアハウスに pull します。
Hail 用の Dataproc Hub 環境を作成する
すでに説明したように、Hail バージョン 0.2.15 の pip インストールにバンドルされているコマンドライン ツール hailctl には、Google Dataproc クラスタで動作する Dataproc というサブモジュールが含まれています。これには、次のコマンドを呼び出すだけで使用できる、完全に構成されたノートブック環境も含まれています。
ただし、Dataproc Hub を使用するなど、Dataproc 向けノートブック機能を活用できるようにするには、Dataproc の初期化操作を行って、Hail で提供されるノートブックが含まれないスタンドアロン バージョンの Hail を使用する必要があります。
Hail を提供する Dataproc クラスタを Dataproc の JupyterLab 環境内に作成するには、次のコマンドを実行します。
クラスタが作成されると緑色のチェックマークで示されます。続いてクラスタ名をクリックし、[ウェブ インターフェース] タブを選択して、JupyterLab のコンポーネント ゲートウェイ リンクをクリックします。

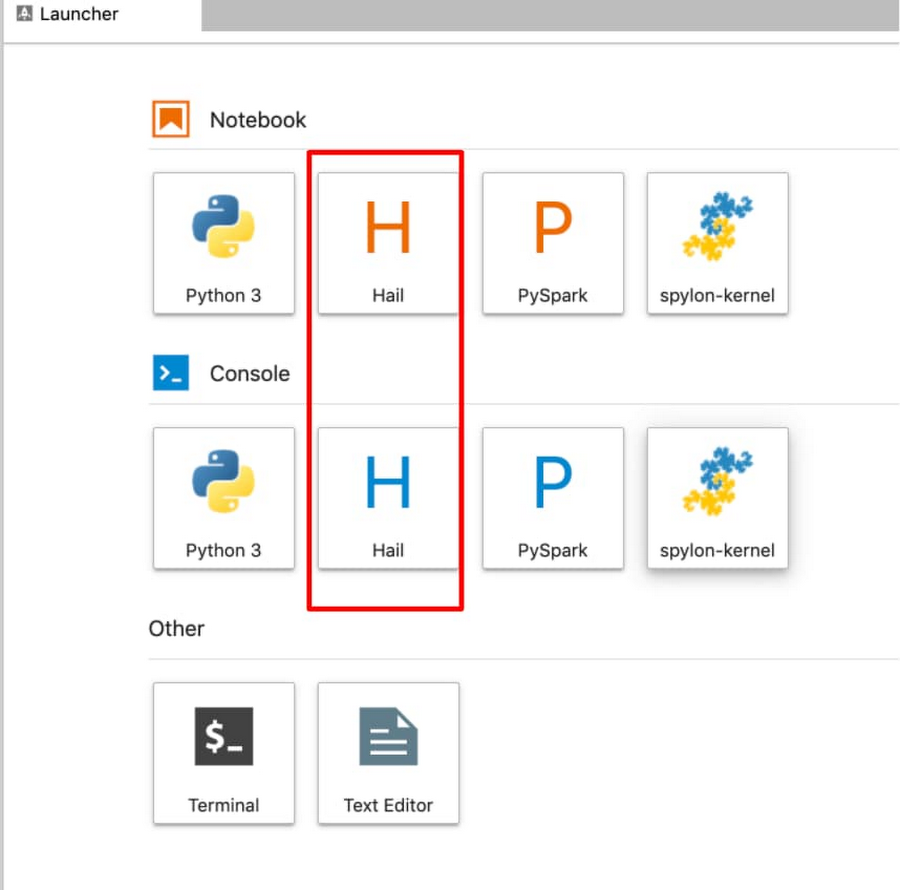
Jupyter IDE 内に、Hail のカーネルとコンソールが表示されます(以下の図の赤枠)。

次に示す実行中のクラスタは、Dataproc クラスタ エクスポート コマンドを実行して簡単に Dataproc Hub 構成に変換できます。
Dataproc Hub と BigQuery を使用してゲノムデータを解析する
Hail for Dataproc で使用できる Jupyter ノートブック環境が構成されました。次に、[分析情報] ゾーンに格納されている BigQuery 遺伝子型データおよび表現型データとのやり取りについて簡単に考察していきましょう。
BigQuery マジックでデータをクエリし、結果を Pandas に返す
SQL ロジックを使って BigQuery 内で GWAS 解析を直接処理し、その処理を BigQuery に push できます。次に、クエリ結果を Pandas データフレームに戻し、可視化してノートブックに表示できます。Dataproc Jupyter ノートブックで、次のように BigQuery マジック コマンドをノートブックのセルに追加するだけで、BigQuery SQL を実行できます。結果は Pandas データフレームに返されます。ノートブックを使って BigQuery で GWAS 解析を行う例は、こちらのチュートリアルでご確認ください。BigQuery の BigQuery ML 機能では、標準 SQL クエリを使って基本的な回帰技術と K 平均法を実施できます。
BigQuery は GWAS や PheWAS の予備的なステップ(特徴量エンジニアリング、データのコホートの定義、記述的分析によるデータの把握)でよく使用されます。BigQuery の一般公開データセットでホストされている 1,000 個のゲノム バリアント データセットを使って、記述的統計情報を見てみましょう。
1000 Genomes Project から、染色体 12 で使用できる SNP データを把握したいとします。Jupyter のセルで、以下をコピーしてセルに貼り付けます。
このクエリにより、my cohort にある 1000 Genomes Project サンプルの基本情報が Pandas データフレームに入力されます。これで、Python と Pandas の標準関数を使用して、このコホートで利用できるデータの検証、プロット、把握を行えるようになりました。
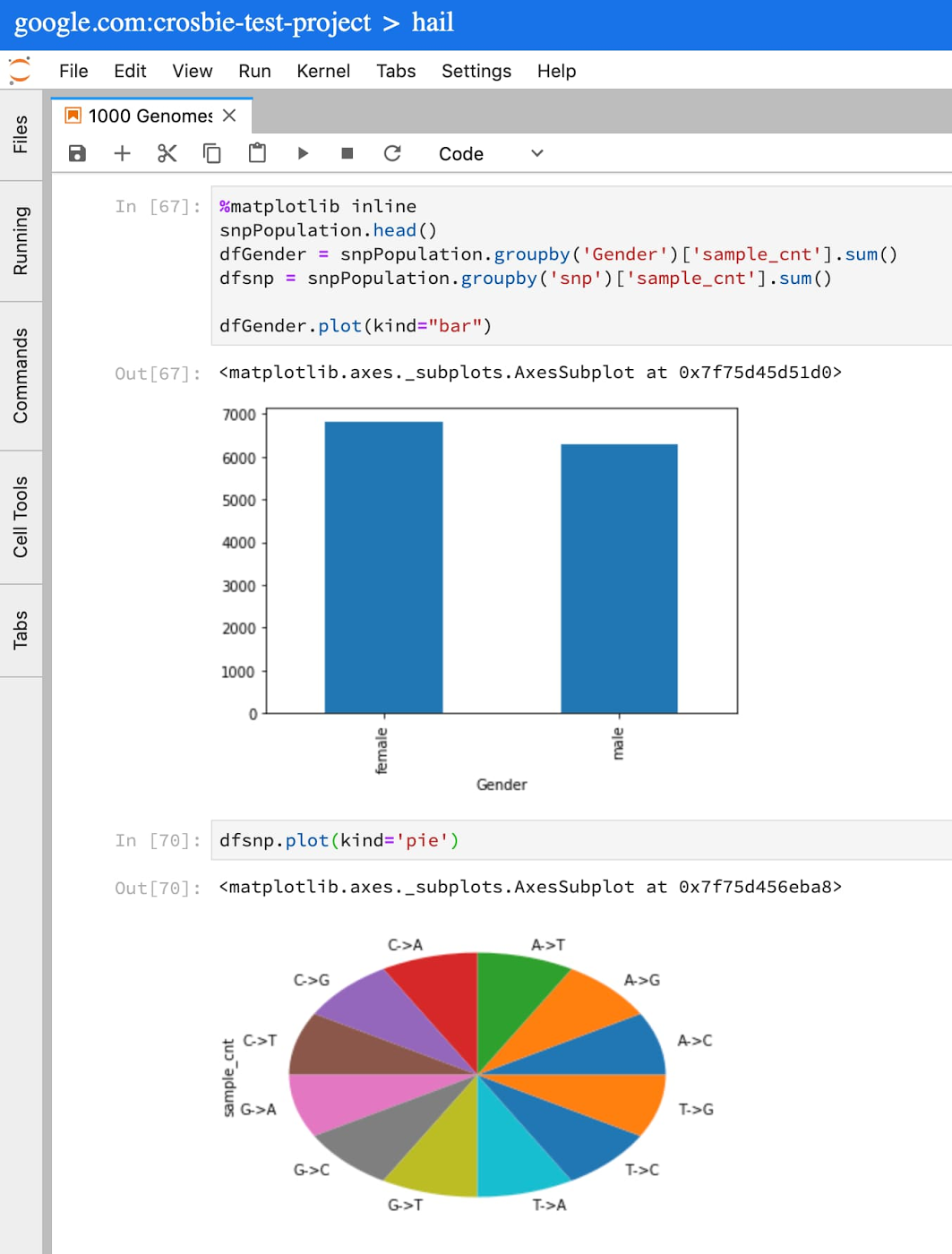

バリアント形式を使用するテーブルに対する SQL クエリの作成について詳しくは、BigQuery を使用したバリアント分析の高度なガイドをご覧ください。
Spark と BigQuery のコネクタを使用して、BigQuery ストレージを Apache Spark 内で直接処理する
集団調査のために大量のゲノムデータを処理する必要があり、Random Forest、Naive Bayes、Gradient Boosted ツリーなどの汎用の分類アルゴリズムと回帰アルゴリズムを使用する場合や、PCA、One Hot Encoding などのアルゴリズムの抽出機能や変換機能を使用する必要がある場合には、Apache Spark で提供する次のような ML 機能を利用できます(ML 機能の一例)。
Apache Spark BigQuery コネクタを Dataproc から使用することで、Apache Spark からデータの読み取りや書き込み行うためのソースとして BigQuery を機能させることができます。この設定方法は、他の Spark データフレームの設定とほぼ同じです。
詳細については、Apache Spark と BigQuery ストレージの統合方法と開始方法をご覧ください。
Variant Transforms ツールを使用して、BigQuery を Hail などのゲノム解析ツール用の VCF に変換する
次のようなゲノムに関するタスクは、Hail によって Spark の基盤の上に提供されるレイヤ(論理層)を使用して行うことができます。
バリアントやサンプルのアノテーションを生成する
トリオ解析によりメンデルの法則の違反を理解する、連鎖不平衡解析によりバリアントを絞り込む、サンプル間で遺伝子相同性を分析する、PCA を使ってサンプルスコアとバリアント荷重を計算する
線形回帰、論理回帰、線形混合回帰を使ってバリアント解析、遺伝的荷重の解析、eQTL 関連分析を行い、遺伝性を予測する
Hail では VCF、BGEN、PLINK で始まるデータ形式を使用します。BigQuery のゲノムデータは、Variant Transforms を使用して BigQuery の VCF 形式から VCF ファイルに簡単に変換できます。VCF を Cloud Storage 上に作成したら、Hail の import_vcf 関数を呼び出します。この関数は、ファイルを変換して Hail の行列表に入力します。
Hail のスケーラブルなゲノム解析の詳細については、Broad Institute の Hail に関する YouTube シリーズをご覧ください。
-プロダクト マネージャー Christopher Crosbie



