Google AI コミュニティによる、クラウドを使用した生物医学研究者の支援
Google Cloud Japan Team
※この投稿は米国時間 2020 年 6 月 27 日に、Google Cloud blog に投稿されたものの抄訳です。
世界的なパンデミックに対応すべく、ホワイトハウスと研究グループの連合体は、世界最大のオンライン データ サイエンス コミュニティ、Kaggle に CORD19 データセットを公開しました。新型コロナウイルスや他の疾患についての理解を深めるという目標は、医療政策、研究、医学界において多くの注目を集めました。Nature の記事によると、Kaggle チャレンジは 3 月中旬に開始されて以来、約 200 万ページビューを獲得しています。
研究者や一般ユーザーが自由に利用できるデータセットには、150,000 以上の学術論文が含まれており、COVID-19(新型コロナウイルス感染症)関連だけで何千にも及びます。そのため、最新の文献の中から、常に最新情報を入手するのは容易ではありません。さらに、COVID-19 やその他の疾患についての科学的理解を深めることができる情報を掲載した医学出版物は数百万にも上ります。そのうえ、こうした文献の多くは機械で容易には利用できず、最新の自然言語処理ツールを使用して要約、分析するのは困難です。
そこで登場したのが Google 人工知能(AI)コミュニティです。これは Machine Learning Google Developer Expert(ML GDE)と呼ばれる社外のデータ サイエンティストのグループで、世界中から選ばれた、極めてスキルの高い AI プラクティショナーのコミュニティです。Google Cloud クレジットのサポートと TensorFlow Research Cloud(TFRC)からのクレジットにより、ML GDE は研究文献の理解の問題に取り組み始めました。彼らは医療の専門家ではありませんが、ビッグデータと AI に関する知識を生物医学の分野に応用することで、現在の危機を支援できるとすぐに察知しました。
4 月に「AI vs COVID-19」(aiscovid19.org)という大胆な名前でチームが結成され、最先端の機械学習とクラウド テクノロジーを利用して、生物医学研究者が研究文献からより迅速に新たな知見を発見できるように支援するという目標を確立しました。
データセットの設計
ML GDE チームが最初に行ったステップは、生物医学研究者に接触して、そのワークフロー、ツール、課題、そして最も重要なこととして医学文献の「関連性」について理解を深めることでした。次のようないくつかの共通の洞察が得られました。
●膨大な量の既存および新しい情報がある
●情報源が不明確で一貫性がない
●現在のツールの情報検索機能に制限がある
○検索が単純なキーワードのみに基づいている
○複数のデータセットが散在している
○文脈に沿った単語の意味を理解できない
現在の AI 革命の特長の一つは、分析するデータが多いほどシステムの能力が向上することです。近年の自然言語処理(BERT、XLNEt、T5、GPT3)では、何百万ものドキュメントを使用して、NLP タスク用の最先端のニューラル ネットワークをトレーニングします。
これらの洞察に基づいて、研究コミュニティを支援する最良の方法は、膨大な論文コーパスを含む 1 つのデータセットを作成し、そのデータセットを機械で使用可能な形式にすることだと判断しました。オープン アクセス運動や Chan Zuckerberg Institute の Meta などのイニシアチブに触発されて、関連性のある、独自の自由に利用できる出版物をできるだけ多く見つけ、AI システムのトレーニングに特化して設計された、簡単にアクセスできる 1 つのデータセットに集約することを目指しました。
BREATHE の紹介
Biomedical Research Extensive Archive To Help Everyone(BREATHE)は大規模な生物医学データベースで、最上位の生物医学研究リポジトリからのエントリを含みます。このデータセットには、英語で公開された 1,600 万を超える生物医学論文のタイトル、要約、本文全文(ライセンスで許可されている場合)が含まれています。2020 年 6 月に最初のバージョンがリリースされました。論文のコーパスは検索クローラによって常に更新されているため、今後新しいバージョンがリリースされる予定です。データセットおよび取得しようとしている分野固有の知識をさらに向上させる方法の一つとして、他の言語(英語以外)で書かれた論文の収集が考えられます。
COVID-19 固有のデータセットはいくつかありますが、BREATHE は次の点で異なります。
●広範 - 多数の異なるソースが含まれる
●機械で読み取り可能
●一般公開されており、自由に使用できる
●スケーラブルで分析しやすく費用対効果の高いデータ ウェアハウス、Google BigQuery でホストされている
BREATHE の開発アプローチ
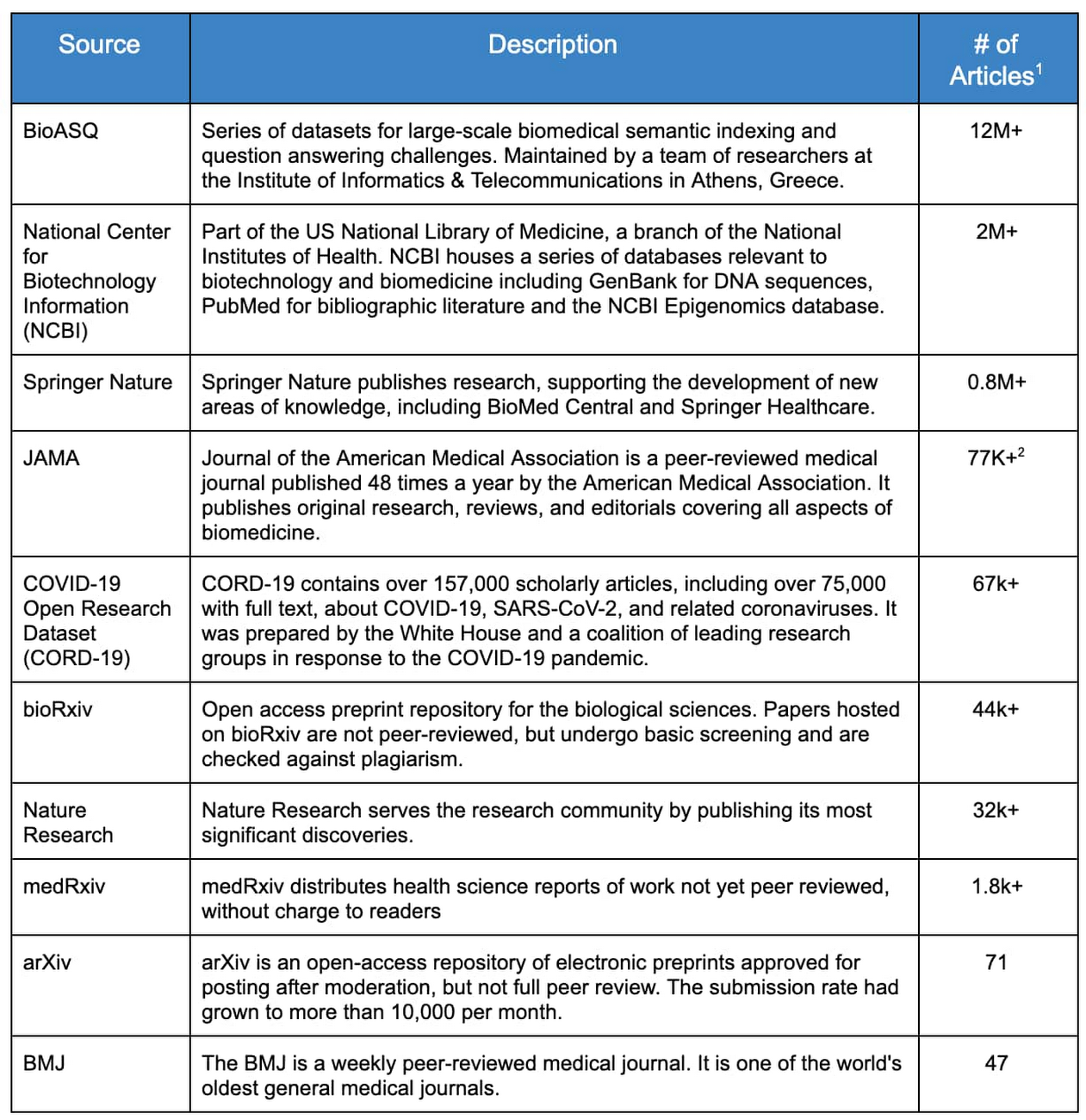
ML GDE チームは、データ量、データ品質、使用可能性という 3 つの主な要素に基づいて、資料が含まれる可能性のある上位 10 のウェブ アーカイブ、つまり「ソース」を特定しました。これらのソースを表 1 に示します。
表 1: 医療アーカイブ

データマイニングのアプローチとツール
論文のダウンロード ワークフローの開発と自動化は、Google Cloud インフラストラクチャを使用することで大幅に加速されました。このシステムは内部では「取り込みパイプライン」と呼ばれており、従来の 3 つの段階、抽出、変換、読み込み(ETL)があります。
Google Cloud Platform での BREATHE データセットの作成
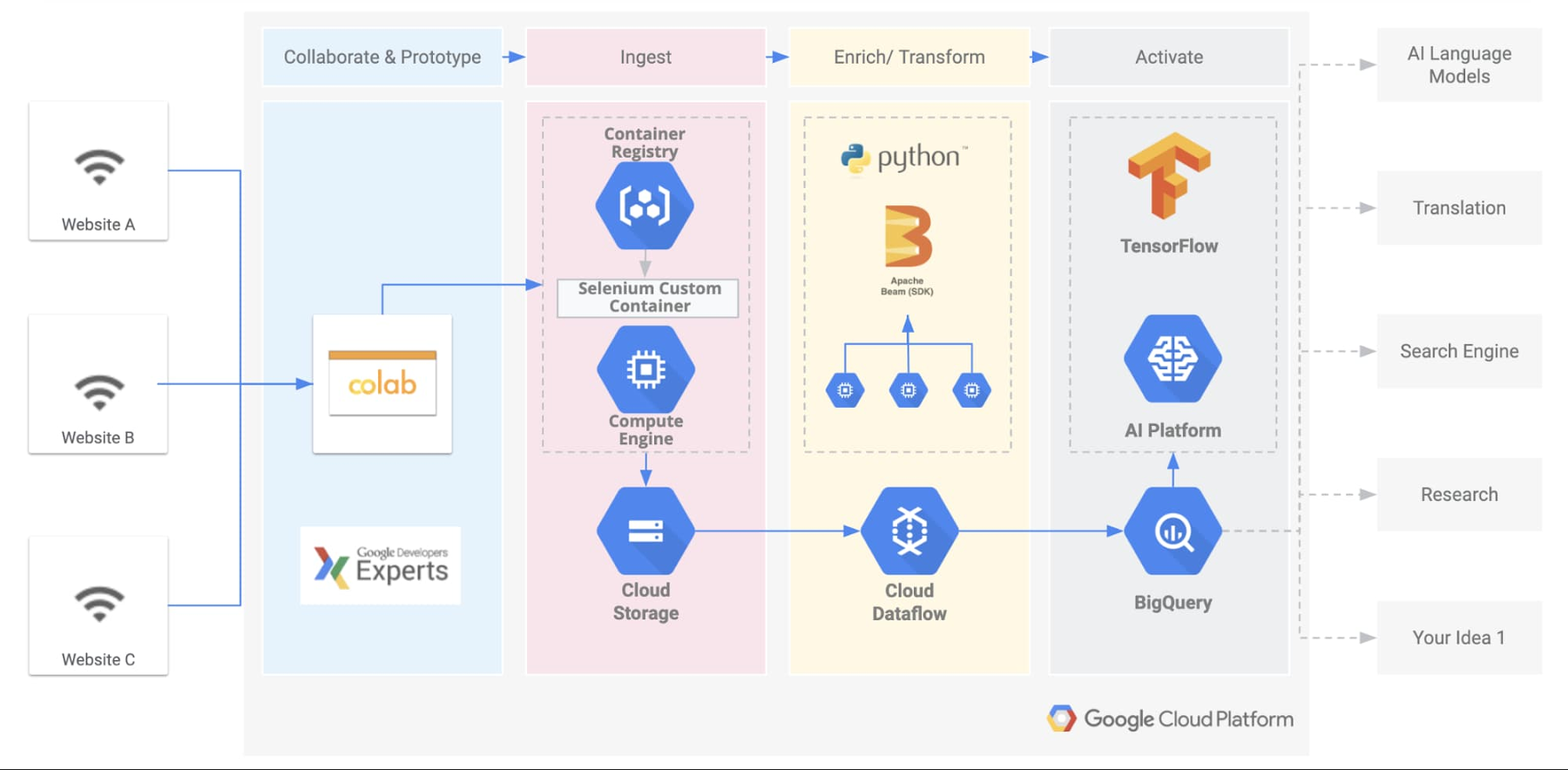

抽出
ML GDE チームは、すべてのリソースについて、まずコンテンツ ライセンスを検証するためにソースの利用規約を遵守していることを確認し、使用可能な場合は API と FTP サーバーを採用しました。残りのリソースについては、一般公開データを取り込むために「倫理的スクレイピング」の理念を導入しました。
スクレーパーのメインロジックのプロトタイプを簡単に作成するために、チームのインターンはGoogle Colaboratory ノートブック(Colab)を使用しました。Colab はホストされている Python Jupyter ノートブックで、これによりユーザーは追加の設定や構成を行わずにブラウザで Python を記述して実行できます。これは時間制限があるものの、GPU に無料でアクセスできるため、多くの機械学習担当者が魅力的なツールとして選択しています。Google Colab を使用することで、インターンや共同作業者の間でコードを簡単に共有することができました。
スクレーパーは Selenium を使用して記述されています。これはウェブブラウザを自動化するためのツールスイートで、その中で Chromium をヘッドレス モードで選択しました(Chromium は Google Chrome ブラウザがベースとしているオープンソース プロジェクトです)。さまざまなソースからすべての元データが Google Cloud Storage バケットに直接ダウンロードされます。
変換
ML GDE チームは、10 の異なるソースから 1,600 万以上の論文を取り込みました。それぞれの論文には、CSV、JSON、または XML 形式の元データと独自のスキーマが含まれています。この膨大な量のデータを効率的に処理するために選択したツールが、Google Dataflow でした。Google Dataflow は、Google Cloud で Apache Beam パイプラインを実行するためのフルマネージド サービスです。変換ステージでは、パイプラインがすべての元ドキュメントを 1 つずつ処理し、クリーニング、正規化、複数のヒューリスティックなルールを適用して、最終的に JSONL 形式の一般的なスキーマを抽出します。適用されるヒューリスティックなルールには、null 値、無効な文字列、重複エントリのチェックが含まれます。また、異なるテーブルにある、異なる名前で同じエンティティを表すフィールド間の一貫性も検証しました。
これらの段階を通過したドキュメントは、操作ステータスに基づいて、最終的に次の 3 つの異なるシンクバケットに入れられます。
●成功: 正しく処理されたドキュメント
●拒否: 1 つ以上のルールに一致しなかったドキュメント
●エラー: パイプラインが処理できなかったドキュメント
Apache Beam を使用すると、単純ではないロジックを読みやすい構文(スニペット 1 のように)で設計できます。Google Dataflow を使用すると、コードを変更することなく、このプロセスを多数の Google Cloud コンピューティング インスタンスの間で簡単にスケールできます。パイプラインは元データ全体に適用され、合計 100 GB の JSONL テキストデータが 1,670 万のレコードに抽出されました。
スニペット 1: Google Dataflow 処理の例
読み込み
最後に、データが Google Cloud Storage バケットと Google BigQuery テーブルに読み込まれました。
BigQuery はインフラストラクチャを管理する必要がなく、データベース管理者も必要ないため、主にデータ サイエンスの専門家で構成されたプロジェクトには最適でした。処理されたドキュメントの総数をスケーリングするため、取り込みプロセスを数回繰り返しました。データ探索の初期段階で、データ サイエンティストは、標準の構造化クエリ言語(SQL)を使用するだけで、BigQuery に読み込まれたデータの内容を調べることができました。
この段階で役立つテクニックの一つは、データセットを「サンプリング」して適合しないドキュメントを発見することです。たとえば、次の単純なコードを使用して、データセット全体の 5% を抽出できます。
より高度なクエリには、Google Colab と BigQuery Python API を使用しました。たとえば、各テーブルの行数を数えるには、以下のようにします。
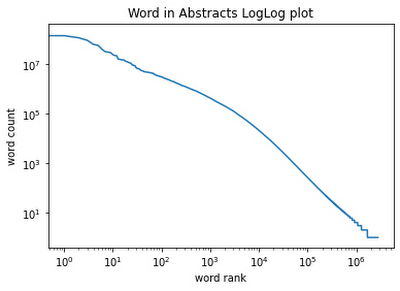
このアプローチを使用して、データセットに関する集計統計を簡単に計算できました。BREATHE 内のすべての要約には、合計 33 億個の単語と 280 万個の一意の単語があります。Python と Colab を使用することで、探索的データ分析も簡単でした。たとえば、次に単語の頻度のプロットを示します。

Google の一般公開データセット プログラム
ML GDE チームは、他のデータ サイエンティストがデータセットの中から価値を見つけ出す可能性があると信じているため、Google の一般公開データセット プログラムを介してデータセットを利用できるようにすることにしました。この公開データセットは Google BigQuery でホストされており、BigQuery の無料枠に含まれています。ユーザーは、それぞれ毎月最大 1 TB まで無料で処理できます。この割り当てにより、誰でも簡単な SQL コマンドを使用して BREATHE データセットを探索できます。こちらの短い動画で BigQuery について学習し、BigQuery の一般公開アクセス プログラムを使用して BREATHE へのクエリを今すぐ始めましょう。
このデータセットでできること
BREATHE データセットをさまざまな方法で使用して、文献の多い生物医学研究について理解を深め、統合し、COVID-19 パンデミックのような生物医学の課題において新たな知見を見いだすことができます。生物医学固有の言語モデルのトレーニング、生物医学情報検索システムの構築、広大な生物医学分野の中のニッチな研究分野に対する新しい形の教師なし分類の導出など、データ サイエンティストが BREATHE を使用して構築できる興味深いものは他にも多数あると ML GDE チームは考えています。たとえば、現在、英語を母国語としない研究者や臨床医は元の著者の言語で資料を理解するしかありませんが、近い将来、論文を多数の異なる言語に正確に翻訳するという困難な課題が解決されるかもしれません。チームでは、AI コミュニティが BREATHE データセットを使用して何を生み出すことができるかを目にするのを楽しみにしています。
クラウド コラボレーション: 力を合わせて
クラウドで作業する明確なメリットの一つは、地理的に離れた場所にいる多数の開発者が 1 つのプロジェクトで共同作業できることです。このケースでは、データセットの生成に 3 つの大陸と 5 つのタイムゾーンの 20 人以上が参画しました。42 Silicon Valley AI および Robotics Lab の責任者である Dan Goncharov 氏が、BREATHE データセット作成の推進チームのリーダーでした。42 は、世界に 16 の拠点を持つ非営利で授業料ゼロの私立のコンピュータ プログラミング学校です。ML GDE チームは、Blaire Hunter 氏、Simon Ewing 氏、Khloe Hou 氏、Gulnozai Khodizoda 氏、Antoine Delorme 氏、Ishmeet Kaur 氏、Suzanne Repellin 氏、Igor Popov 氏、Uliana Popova 氏の協力、特に Ivan Kozlov 氏、Francesco Mosconi 氏(ゼロからディープ ラーニングまで)、Fabrizio Milo 氏(エントロピー ソース)の協力に感謝の意を表します。
次回: TensorFlow と最先端の自然言語アーキテクチャを使用した検索ツールの構築
この投稿では、プロジェクトのバックグラウンド、設計の原則、そして生物医学研究者向けに公開された、機械で読み取り可能なデータセットである BREATHE を作成するための開発プロセスについて説明しました。次の投稿では、ML GDE チームが、オープンソースと最先端の自然言語理解ツールを使用して、このデータセット上に単純な検索ツールを構築した様子を紹介します。
BREATHE の作成に使用したツール
●Google ネットワーキングとコンピューティング
●Google Dataflow
●Google BigQuery(BQ)
●Google Cloud Storage(GCS)
●Google Cloud 一般公開データセット プログラム
●Selenium
●Google Colab
●Python 3
1. 「独自」の論文とは、DOI によって判断されますが、同じ DOI でリストされる論文の多くにも貴重な追加情報が含まれています。
2. JAMA には、7 万個以上の論文の本文全体が、他のソースから抽象的な形式で技術的に複製されて含まれています。



